Sumário
- Materiais para este módulo
- Apresentação do curso
- Instalação do R e RStudio para o SO Windows
- Como utilizar o R e o RStudio
- História do R
- Como o R trabalha
- Comandos no R e Ambiente Global
- Arquivos
.Rdatae.Rhistory - Criando e salvando um script
- Objetos
- Vetores
- Matrizes bidimensionais
- Matrizes multidimencionais
- Listas de dados
- Quadro de dados
- Importando dados
- Funções
- Boas práticas de como escrever um código
- Pacotes
- Ambientes e Caminho de busca
- Interfaces com outras linguagens
- Considerações e preparação para o módulo Programação em R (Nível Intermediário)
Slides de Aulas
- Aula 00: Apresentação do Curso
- Aula 01: Instalação do R e do RStudio
- Aula 02: Passos iniciais para a utilização do R e do RStudio
- Aula 03: História do R
- Aula 04: Como o R trabalha
- Aula 05: Comandos no R e Ambiente Global
- Aula 06: Arquivos
.RDatae.Rhistory - Aula 07: Criando e salvando um Script
- Aula 08: Objetos
- Aula 09: Vetores
- Aula 10: Matrizes
- Aula 11: Arrays
- Aula 12: Lists
- Aula 13: Data frames
- Aula 14: Importando dados
- Aula 15: Funções
- Aula 16: Estruturas de controle
- Aula 17: Como criar funções
- Aula 18: Boas práticas de como escrever um código
- Aula 19: Pacotes
- Aula 20: Carregando e Anexando pacotes
- Aula 21: NAMESPACE de um pacote
- Aula 22: Quando devemos utilizar os operadores
::e::: - Aula 23: Ambientes e Caminhos de busca
- Aula 24: Interface com outras linguagens
- Aula 25: Considerações e Preparação para a Programação em R (Nível Intermediário)
Scripts
- Script 00: ~
- Script 01: ~
- Script 02: ~
- Script 03: Escopo léxico
- Script 04: ~
- Script 05: Comandos
- Script 06: ~
- Script 07: .RData e .Rhistory
- Script 08: Script08.txt; Script 08_2: Script08_2.txt
- Script 09: Vetores
- Script 10: Matrizes bidimensionais
- Script 11: Matrizes multidimensionais
- Script 12:
- Script 13:
- Script 14: alfafa.txt, datast1980.txt, producao.csv
- Script 15:
- Script 16:
- Script 17:
- Script 18:
- Script 19: Script19.txt
- Script 20: Script20.txt
- Script 21: Script21.txt
- Script 22: Script22.txt
- Script 23: Script23.txt
- Script 24: r-python.R, r-rcpp.R, r-tcltk.R, add.cpp, add.py
- Script 25: ~
Exercícios e Scripts via Shiny
Apresentação do curso
O Curso R será todo aprensentado no formato online, sendo que as aulas terão vídeos como suporte, postados no canal Youtube/Ben Dêivide divulgados ao longo do material. Os vídeos serão bem objetivos de curta duração para que usem como suporte com o material escrito. Como complemento desse material, disponibilizaremos os scripts com os comandos utilizados em cada aula e sua versão em Shiny para os que não quiserem realizar inicialmente a instalação do R e do RStudio, poderão utilizar uma versão online do material juntamente com a linguagem R.
Instalação do R e RStudio para o SO Windows
A linguagem R é o objetivo principal desse curso. Assim, faremos inicialmente a sua instalação, uma vez que o RStudio é apenas uma GUI, e sem o R, não há sentido instalá-lo.
Dessa forma, seguem os passos para a instalação:
- Instalação do
R: https://cran.r-project.org/bin/windows/base/ - Instalação do RStudio: https://rstudio.com/products/rstudio/download/#download
Justificamos a utilizamos do RStudio, pela quantidade de recursos disponíveis e a diversidade de usuários R, que hoje o perfil não é apenas de um programador, mas um usuário que necessita de uma ferramenta estatística para análise de seus dados. Dessa forma, até por questão de praticidade, e de uso pessoal, não deixaremos de repassar o entendimento sobre a linguagem R com o uso do RStudio.
Como utilizar o R e o RStudio
A primeira ideia que temos sobre a linguagem R é a linha de comando no R, que é simbolizada pelo prompt de comando “>.” Este símbolo significa que o R está pronto para receber os comandos do usuário. O prompt de comando está localizado no console do R. Vejamos o console do R a seguir, que é o local que recebe as linhas de comando do usuário.
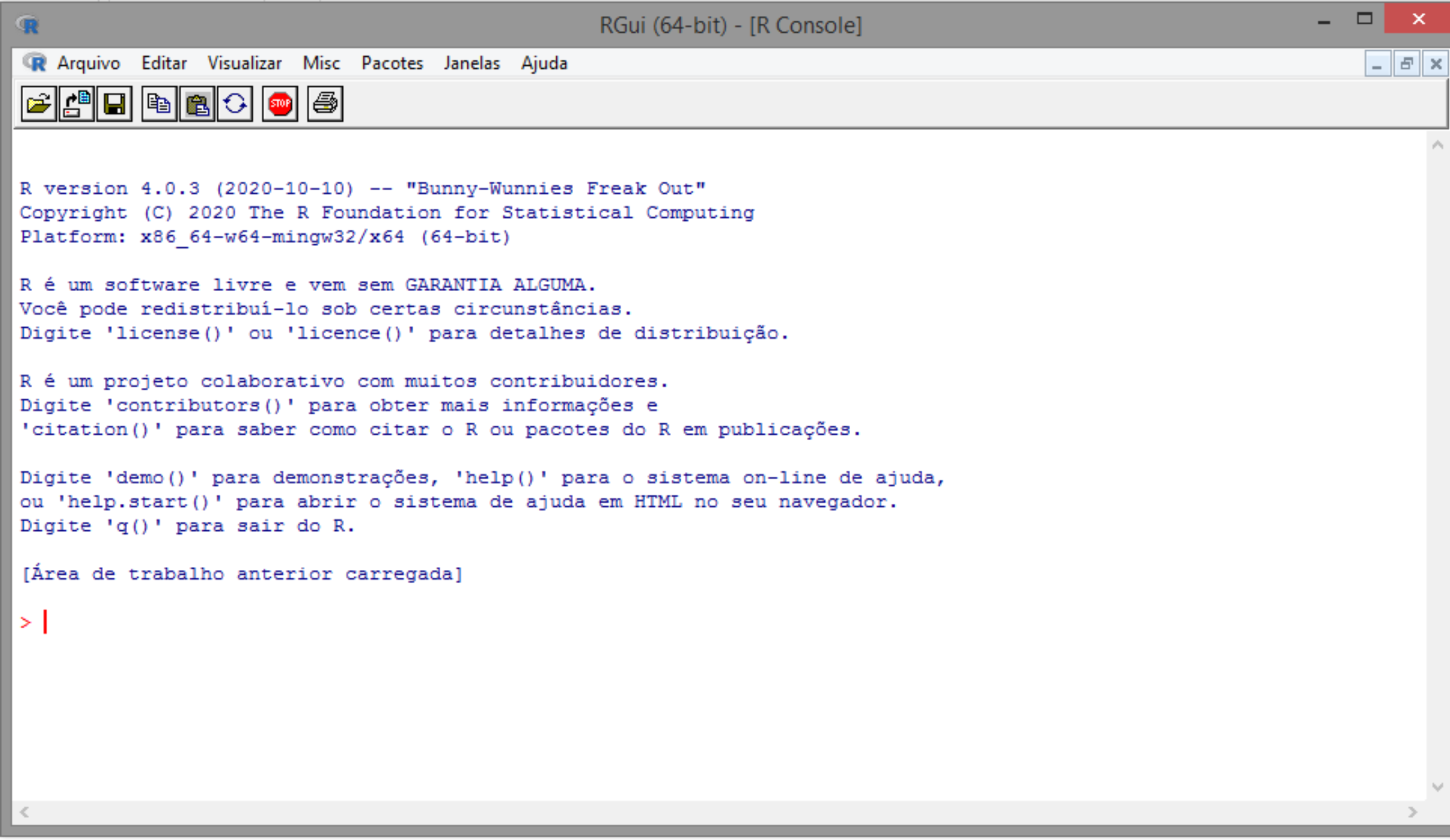
Figure 1: Console do R (Versão 4.0.3).
O R ao ser iniciado está pronto para ser inserido as linhas de comando desejadas. Uma forma simples de armazernar os seus comandos é por meio de um script, isto é, um arquivo de texto com extensão .R. Para criar basta ir em: Arquivo > Novo script.... Muitas outras informações iremos ver ao longo do curso.
O RStudio se apresenta como uma interface para facilitar a utilização do R, tendo por padrão quatro quadrantes, apresentados na Figura 2.
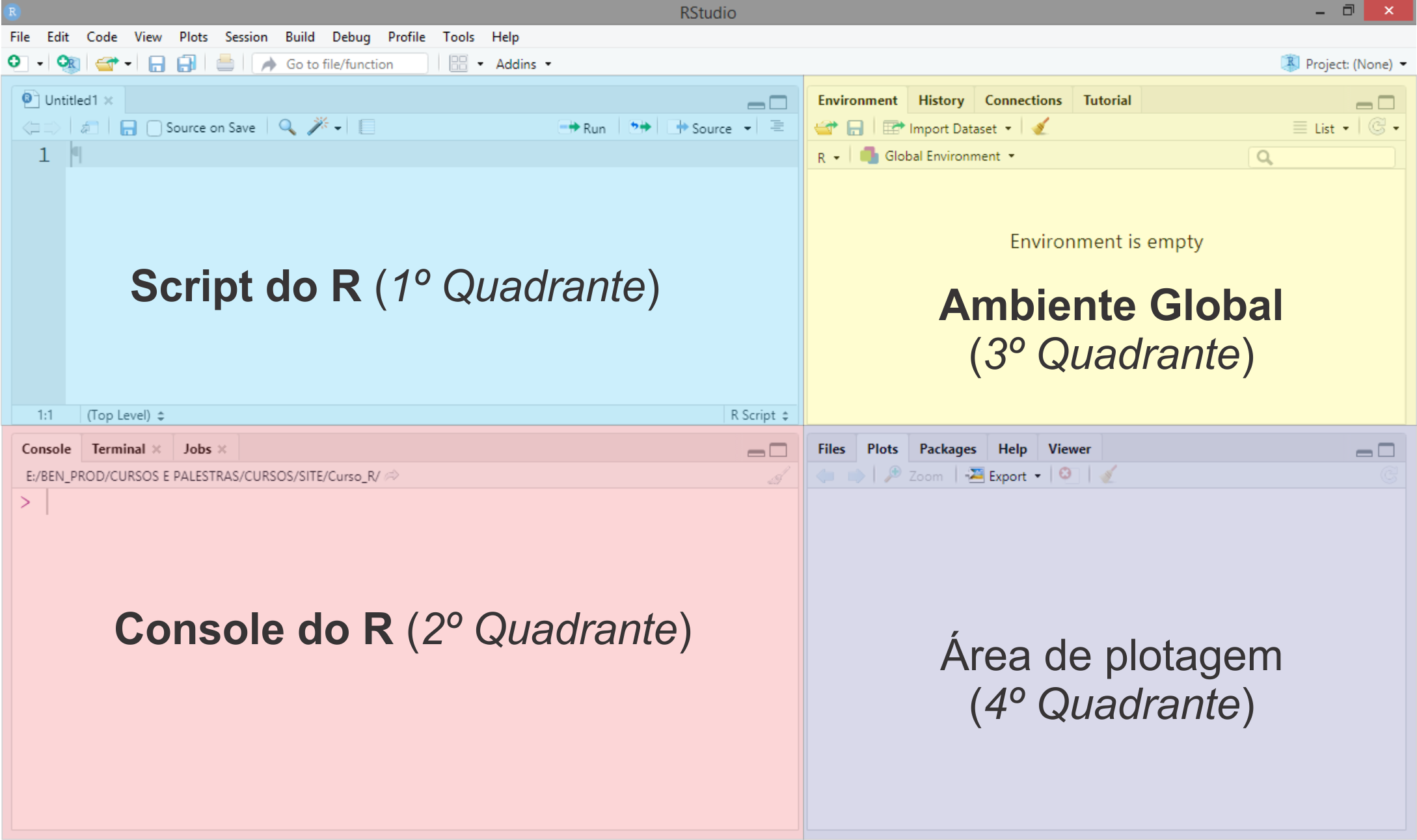
Figure 2: Interface do RStudio (Versão 1.4.1103).
Muitas coisas na interface do R podem se tornar problemas para os usuários, uma vez que janelas gráficas, janelas de scripts, dentre outras, se sobrepõe. Uma vantagem no RStudio foi essa divisão de quadrantes, que torna muito mais organizado as atividades realizadas no R. De um modo geral, diremos que o primeiro quadrante é responsável pela entrada de dados, comandos, isto é, o input. O segundo quadrante, que é o console do R, representa tanto entrada como saída de informações (input/output). Dependendo as atividades as abas podem aumentar. O terceiro quadrante representa informações básicas como objetos no ambiente global, a memória de comandos na aba History, dentre outras, e também representa entrada como saída de informações (input/output). Por fim, o quarto quadrante é responsável por representação gráficas, instalação de pacotes, renderização de páginas web.
História do R
A linguagem R tem a sua primeira aparição científica publicada em 1996, com o artigo intitulado R: A Language for Data Analysis and Graphics, cujos os autores são os desenvolvedores da linguagem, George Ross Ihaka e Robert Clifford Gentleman.
![Criadores do R.^[Fonte das fotos: Robert Gentleman do site: https://biocasia2020.bioconductor.org/ e Ross Ihaka do site: https://www.stat.auckland.ac.nz/en/about/news-and-events-5/news/news-2017/2017/12/ross-ihaka-retires.html]](images/criadores_r.png)
Figure 3: Criadores do R.1
Durante a época em que estes professores trabalhavam na Universidade de Auckland, Nova Zelândia, desenvolvendo uma implementação alternativa da lingugagem S, desenvolvida por John Chambers, que comercialmente era o S-PLUS, nasceu em 1991, o projeto da linguagem R, em que em 1993 o projeto é divulgado e em 1995, o primeiro lançamento oficial, como software livre com a licença GNU. Devido a demanda de correções da linguagem que estava acima da capacidade de atualização em tempo real, foi criado em 1997, um grupo central voluntário, responsável por essas atualizações, o conhecido R Development Core Team2, que hoje está em 20 membros (atualizado em 09 novembro, 2021): Douglas Bates, John Chambers, Peter Delgaard, Robert Gentleman, Kurt Hornik, Ross Ihaka, Tomas Kalibera, Michael Lawrence, Friedrich Leisch, Uwe Ligges, Thomas Lumley, Martin Maechler, Sebastian Meyer, Paul Murrel, Martyn Plummer, Brian Ripley, Deepayan Sarkarm, Duncan Temple Lang, Luke Tierney e Simon Urbanek.
Por fim, o CRAN (Comprehensive R Archive Network) foi oficialmente anunciado em 23 de abril de 19973. O CRAN é um conjunto de sites (espelhos) que transportam material idêntico, com as contribuições do R de uma forma geral.
R é uma linguagem de programação e ambiente de software livre e código aberto (open source). Entendemos4:
- Software livre: software que respeita a liberdade e sendo de comunidade dos usuários, isto é, os usuários possuem a liberdade de executar, copiar, distribuir, estudar, mudar, melhorar o software. Ainda reforça que um software é livre se os seus usuários possuem quatro liberdades:
- Liberdade 0 - A liberdade de executar o programa como você desejar, para qualquer propósito;
- Liberdade 1 - A liberdade de estudar como o programa funciona, e adaptá-la as suas necessidades;
- Liberdade 2 - A liberdade de redistribuir cópias de modo que você possa ajudar outros;
- Liberdade 3 - A liberdade de distribuir cópias de suas versões modificadas a outros.
Algo que deve está claro é que um software livre não significa não comercial. Sem esse fim, o software livre não atingiria seus objetivos.
Agora perceba que, segundo Richard Stallman5, a ideia de software livre faz campanha pela liberdade para os usuários da computação. Por outro lado, o código aberto valoriza principalmente a vantagem prática e não faz campanha por princípios.
- Código aberto: Para Richard Stallman6 código aberto apoia critérios um pouco mais flexíveis que os do software livre. Todos os códigos abertos de software livre lançados se qualificariam como código aberto. Quase todos os softwares de código aberto são software livre, mas há exceções, como algumas licenças de código aberto que são restritivas demais, de forma que elas não se qualificam como licenças livres. Nesse contexto, o autor cita muitas situações que diferenciam os dois termos. Vale a pena a leitura.
A linguagem R é uma combinação da linguagem S com a semântica de escopo léxico da linguagem Scheme. Dessa forma, a linguagem R se diferencia em dois aspectos principais7:
- Gerenciamento de memória: usando as próprias palavras de Ross Ihaka8, em R, alocamos uma quantidade fixa de memória na inicialização e a gerenciamos com um coletor de lixo dinâmico. Isso significa que há muito pouco crescimento de heap e, como resultado, há menos problemas de paginação do que os vistos na linguagem S.
- Escopo: na linguagem
R, as funções acessam as variáveis criadas no corpo da própria função, como também as variáveis contidas no ambiente que a função foi criada. No caso da linguagemS, isso não ocorre, assim, como por exemplo na linguagemC, em que as funções acessam apenas variáveis definidas globalmente.
Vejamos alguns exemplos para entendimento (Se você ainda não está ambientado ao R, estude esse módulo primeiro, e depois reflita sobre esses exemplos). Antes de executar as linhas de comando, instale o pacote lobstr como segue:
# Instale o pacote lobstr
install.packages("lobstr")
- Exemplo 1: As funções têm acesso ao escopo em que foram criadas.
# Criando um nome "n" associado a um objeto 10 no escopo da funcao
n <- 10
# Criando um nome "funcao" associado a um objeto que eh uma funcao
funcao <- function() {
print(n)
}
# Imprimindo 'funcao'
funcao()
[1] 10- Exemplo 2: As variáveis criadas ou alteradas dentro de uma função, permanecem na função.
# Criando um nome "n" associado a um objeto 10 no escopo da funcao
n <- 10
lobstr::obj_addr(n) # Identificador do objeto
[1] "0x2ad506d0"# Criando um nome "funcao" associado a um objeto que eh uma funcao
funcao <- function() {
# Imprimindo n
print(n)
# Criando um nome "n" associado a um objeto 15 no corpo da funcao
n <- 15
# Imprimindo n
print(n)
}
# Imprimindo 'funcao'
funcao()
[1] 10
[1] 15# Imprimindo 'n'
n
[1] 10lobstr::obj_addr(n) # Identificador do objeto
[1] "0x2ad506d0"- Exemplo 3: As variáveis dentro de uma função permanecem nelas, exceto no caso em que a atribuição ao escopo seja explicitamente solicitada.
# Criando um nome "n" associado a um objeto 10 no escopo da funcao
n <- 10
lobstr::obj_addr(n) # Identificador do objeto
[1] "0x287b2b18"# Criando um nome "funcao" associado a um objeto que eh uma funcao
funcao <- function() {
# Imprimindo n
print(n)
# Criando um nome "n" associado a um objeto 15 no corpo da funcao
n <<- 15
# Imprimindo n
print(n)
}
# Imprimindo 'funcao'
funcao()
[1] 10
[1] 15# Observe que depois de usar a superatribuicao ("<<-") dentro da funcao,
#o nome "n" passou a estar associado ao numero 15 e nao mais ao numero 10, observe
n
[1] 15lobstr::obj_addr(n) # Identificador do objeto
[1] "0x287b2a38"- Exemplo 4: Por fim, embora a linguagem
Rtenha um escopo padrão, chamado ambiente global, os escopos de funções podem ser alterados.
# Criando um nome 'n' associado a um objeto 10 no escopo da funcao (ambiente global)
n <- 10
# Criando um nome 'funcao' associado a um objeto que eh uma função criado no ambiete global
funcao <- function() {
# Imprimindo n
print(n)
}
# Imprimindo 'funcao' no ambiente global
funcao()
[1] 10# Criando um novo ambiente
novo_ambiente <- new.env()
# Criando um nome "n" associado ao objeto 20 no ambiente 'novo_ambiente'
novo_ambiente$n <- 20
# Criando um objeto funcao no ambiente 'novo_ambiente'
environment(funcao) <- novo_ambiente
# Imprimindo 'funcao' no ambiente 'novo_ambiente'
funcao()
[1] 20Como a linguagem S é também uma linguagem interpretada cuja base é a linguagem FORTRAN, a linguagem R também é uma linguagem interpretada e baseada além da linguagem S, tem como base as linguagens de baixo nível C e FORTRAN e a própria linguagem R.
Embora o R tenha uma interface baseada em linhas de comando, existem muitas interfaces gráficas ao usuário com destaque ao RStudio, criado por Joseph J. Allaire, Figura 4.
![J. J. Allaire, o criador do RStudio^[Fonte da foto: https://rstudio.com/speakers/j.j.-allaire/].](images/jjallaire.jpg)
Figure 4: J. J. Allaire, o criador do RStudio9.
Essa interface tornou o R mais popular, pois além de produzir pacotes de grande utilização hoje como a família de pacotes tidyverse, rmarkdown, shiny, dentre outros, permite uma eficiente capacidade de trabalho de análise de utilização do R. Uma vez que o RStudio facilita a utilização de muitos recursos por meio de botões, como por exemplo, a criação de um pacote R. A quem diga que para um iniciante em R, não seja recomendado utilizar o RStudio para o entendimento da linguagem. Cremos, que o problema não é a IDE10 utilizada, e sim, o caminho onde deseja chegar com a linguagem R.
No Brasil, o primeiro espelho do CRAN foi criado na UFPR, pelo grupo do Prof. Paulo Justiniano. Inclusive um dos primeiros materiais mais completos sobre a linguagem R produzidos no Brasil, foi dele, iniciado em 2005, intitulado Introdução ao Ambiente Estatístico R. Vale a pena assistirmos o evento a palestra: R Releflões: um pouco de história e experiências com o R, proferida pelo Prof. Paulo Justiniano Ribeiro Júnior, no R Day - Encontro nacional de usuários do R, ocorrido em 2018 em Curitiba/UFPR, do qual o vídeo está disponível no Canal (Youtube) LEG UFPR.
Para quem não sabe, o Prof. Paulo Justiniano (Figura 5) e sua equipe trouxeram o primeiro espelho do R para o Brasil e foi o desenvolvedor de um dos 100 primeiros pacotes submetidos ao CRAN11. Devido a sua ida para Lancaster/Inglaterra para fazer o doutorado, em 1997, se deparou com o início dos estudos sobre o R fora do país. Isso porque havia uma necessidade muito grande de um programa para ser utilizado nas plataforma UNIX. Nesse momento a linguagem S estava sendo uma saída para isso, porém, o uso de memória que a linguagem consumia nos computadores, na criação das rotinas, era muito grande (Claro que grande para aquela época!). Não satisfeito, ao voltar para o Brasil, em 2002, se depara com alguns usuários da linguagem S, porém, certo como mudança de cultura na utilização de linguagem R, passou a densevolver materiais didáticos da linguagem, para as disciplinas do departamento de Estatística (UFPR).
![Foto de divulgação de Paulo Justiniano^[Fonte da foto: https://www.ufpr.br/portalufpr/noticias/disciplinas-transversais-para-programas-de-pos-graduacao-abrem-inscricoes-nesta-segunda-feira/] no *R Day*.](images/paulo_justiniano.jpeg)
Figure 5: Foto de divulgação de Paulo Justiniano12 no R Day.
Como o R trabalha
Iniciamos a discussão por uma afirmação de John McKinley Chambers, do qual afirmou que o R tem três princípios (Chambers 2016):
], o criador da linguagem `S`.](images/john_chambers.jpg)
Figure 6: John Chambers13, o criador da linguagem S.
- Princípio do Objeto: Tudo que existe em
Ré um objeto;- Princípio da Função: Tudo que acontece no
Ré uma chamada de função;- Princípio da Interface: Interfaces para outros programas são parte do
R.
Ao longo de todo o curso, para os três módulos, iremos nos referir a esses princípios. Vamos inicialmente observar uma adaptação da ilustração feita por Paradis (2005), mostrando como o R trabalha, Figura 7.
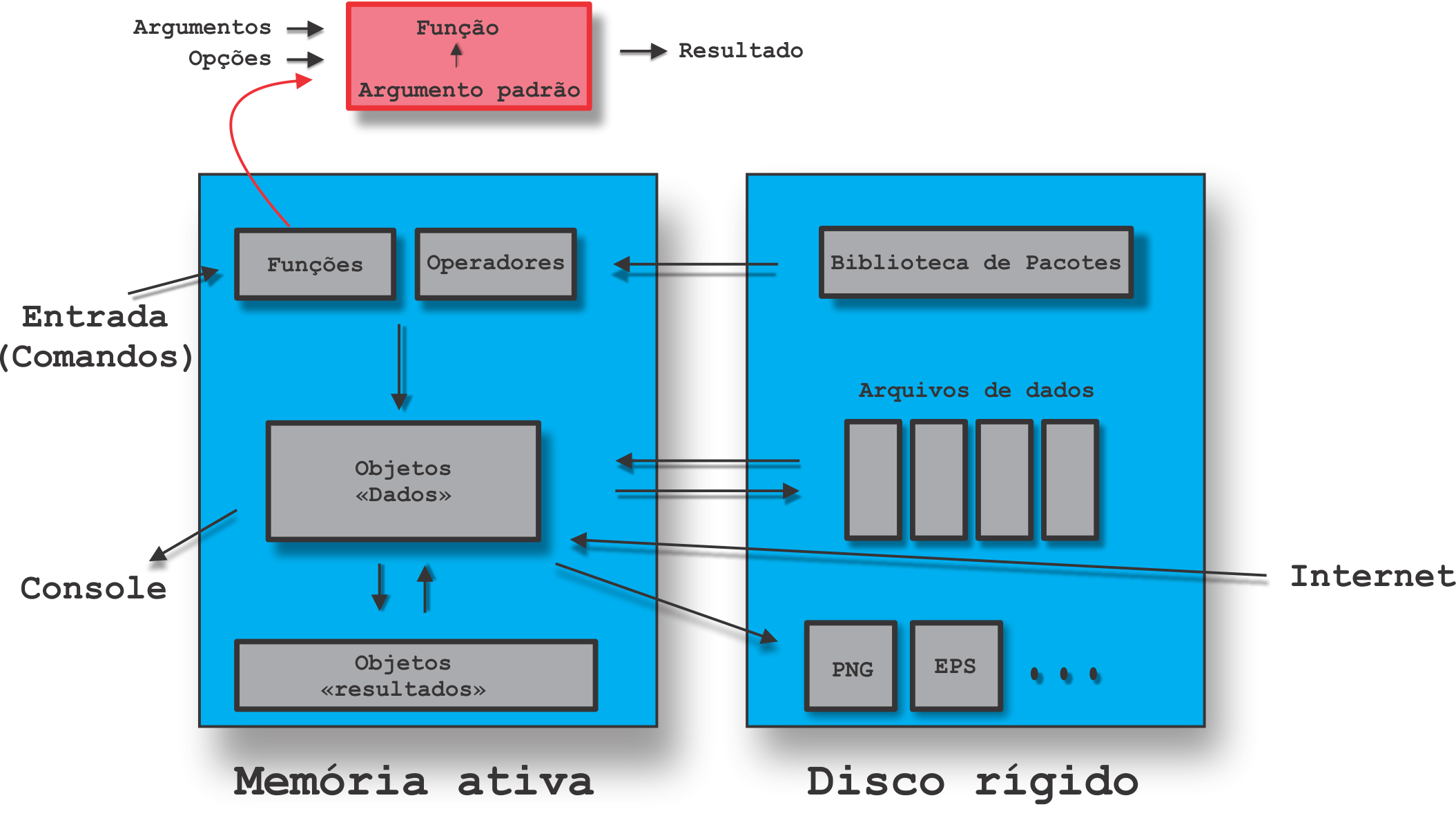
Figure 7: Esquema de como o R funciona.
Toda ação que acontece no R é uma chamada de função (Operadores e funções), que por sua vez é armazenada na forma de um objeto, e este se associa a um nome. A forma de execução de uma função é baseada em argumentos (dados, fórmulas, expressões, etc), que são entradas, ou argumentos padrões que já são pré-estabelecidos na criação da função. Esses tipos de argumentos podem ser modificados na execução da função. Por fim, a saída é o resultado, que é também um objeto, e pode ser usado como argumento de outras funções.
Na Figura 7, observamos que todas as ações realizadas sobre os objetos ficam armazenadas na memória ativa do computador. Esses objetos são criados por comandos (teclado ou mouse) através de funções ou operadores (chamada de função), dos quais leem ou escrevem arquivo de dados do disco rígido, ou leem da própria internet. Por fim, o resultado desses objetos podem ser apresentados no console (memória ativa), exportados em formato de imagem, página web, etc. (disco rígido), ou até mesmo ser reaproveitado como argumento de outras funções, porque o resultado também é um objeto.
Comandos no R e Ambiente Global
Console e Prompt de comando
Como falado anteriormente, o R é uma linguagem baseada em linhas de comando, e as linhas de comando, são executadas uma de cada vez, no console. Assim que o prompt de comando está visível na tela do console, o R indica que o usuário está pronto para inserir as linhas de comando. O símbolo padrão do prompt de comando é “>,” porém ele pode ser alterado. Para isso, use a linha de comando, por exemplo:
options(prompt = "R>")
# Toda vez que o console iniciar, começarar por 'R>'
10
[1] 10O conjunto de símbolos que podem ser utilizados no R depende do sistema operacional e do país em que o R está sendo executado. Basicamente, todos os símbolos alfanuméricos podem ser utilizados, mas para evitar problemas quanto ao uso das letras aos nomes, opte pelos caracteres ASCII.
A escolha do nome associado a um objeto tem algumas regras:
- Deve consistir em letras, dígitos,
.e_; - Os nomes devem ser iniciado por uma letra ou um ponto não seguido de um número, isto é, Ex.:
.123,1n, dentre outros; - As letras maiúsculas se distinguem das letras minúsculas;
- Não pode inicia por
_ou dígito, é retornado um erro no console caso isso ocorra; - Não pode usar qualquer uma das palavras reservadas pela linguagem, isto é,
TRUE,FALSE,if,for, dentre outras, que pode ser consultado usando o comando?Reserved().
Um nome que não segue essas regras é chamado de um nome não sintático. Um comando que pode ser usado para converter nomes não sintatícos em nomes sintáticos é make.names.
Apesar dessas justificativas, algumas situações como as apresentadas nos exemplos anteriores são possíveis, ver Wickham (2019) na Seção 2.2.1
Comandos elementares
Os comandos elementares podem ser divididos em expressões e atribuições. Por exemplo, podemos está interessados em resolver a seguinte expressão \(10 + 15 = 25\). No console quando passamos pelo comando:
10 + 15
[1] 25O R avalia essa expressão internamente e imprime o resultado na tela, após apertar o botão ENTER do teclado. Esse fato é o que ocorre no segundo princípio mencionado por Chambers (2016), tudo em R acontece por uma chamada de função. Na realidade o símbolo + é uma função interna do R, que chamamos de função primitiva, porque foi implementada em outra linguagem. Assim, essa é o resultado de três objetos (“10,” “+,” “15”) que são avaliados internamente, do qual a função + é chamada, e em seguida o resuldo é impresso no console.
Do mesmo modo, se houver algum problema em algum dos objetos o retorno da avaliação pode ser uma mensagem de erro. Um caso muito prático é quando utilizamos o separador de casas decimais para os números sendo a vírgula. Quando na realidade deve ser um ponto “.” respeitando o sistema internacional de medidas. A vírgula é utilizada para separar elementos, argumentos em uma função, etc. Vejamos o exemplo:
10,5 + 15,5Error: <text>:1:3: ',' inesperado
1: 10,
^Porém, tem que ficar claro que uma expressão é qualquer comando repassado no console. Este comando é avaliado e seu resultado impresso, há menos que explicitamente o usuário queira torná-lo invisível14. Caso algum elemento do comando não seja reconhecido pelo R, há um retorno de alguma mensagem em forma de “erro” ou “alerta,” tentando indicar o possível problema. Todos esses processos ocorrem na memória ativa do computador, e uma vez o resultado impresso no console, o valor é perdido, há menos que você atribua essa expressão a um nome, que erroneamento usamos o termo: “criamos um objeto!” A atribuição dessa expressão será dada pela junção de dois símbolos “<-,” falado mais a frente. Um comando em forma de atribuição também avalia a sua expressão, um nome se associa ao seu resultado, e o resultado será mostrado, se posteriormente, após a execução você digitar o “nome” atribuído a esse resultado. Vejamos um exemplo:
# Foi criado um objeto do tipo caractere e o nome "meu_nome" foi associado a ele
# O 'R' avalia essa expressão, mas não imprime no console!
meu_nome <- "Ben"
# Para imprimir o resultado da expressão, digitamos o nome "meu_nome" no console
# e apertamos o botão ENTER do teclado!
meu_nome
[1] "Ben"Execução dos comandos
Quando inserimos um comando no console, executamos uma linha de comando por vez ou separados por “;” em uma mesma linha. Vejamos
# Uma linha de comando por vez
meu_nome <- "Ben" # Criamos e associamos um nome ao objeto
meu_nome # Imprimos o objeto
[1] "Ben"# Tudo em uma linha de comando
meu_nome <- "Ben"; meu_nome
[1] "Ben"Se um comando for muito grande e não couber em uma linha, ou caso deseje completar um comando em mais de uma linha, após a primeira linha haverá o símbolo “+” iniciando a linha seguinte ao invés do símbolo de prompt de comando (“>”), até que o comando esteja sintaticamente completo. Vejamos:
# Uma linha de comando em mais de uma linha
(10 + 10) /
2
[1] 10Por fim, todas linhas de comando quando iniciam pelo símbolo jogo da velha, “#” indica um comentário e essa linha de comando não é avaliada pelo console, apenas impressa na tela. E ainda, as linhas de comandos no console são limitadas a aproximadamente 4095 bytes (não caracteres).
Chamada e correção de comandos anteriores
Uma vez que um comando foi executado no console, esse comando por ser recuperado usando as teclas de setas para cima e para baixo do teclado, recuperando os comandos anteriorermente executados, e que os caracteres podem ser alterados usando as teclas esquerda e direita do teclado, removidas com o botão Delete ou Backspace do teclado, ou acrescentadas digitando os caracteres necessários. Uma outra forma de completar determinados comandos já existentes, como por exemplo, uma função que já existe nas bibliotecas de instalação do R, usando o botão Tab do teclado. O usuário começa digitando as iniciais, e para completar o nome aperta a tecla Tab. Posteriormente, basta completar a linha de comando e apertar ENTER para executá-la. Vejamos um exemplo a seguir.
Usando esses recursos no RStudio são mais dinâmicos e vão mais além. Por exemplo, quando usamos um objeto do tipo função, estes apresentam o que chamamos de argumento(s) dentro do parêntese de uma função, do qual são elementos necessários, para que a função seja executada corretamente. Nesse caso, ao inseri o nome dessas funções no console, usando o RStudio, ao iniciá-la com a abertura do parêntese, abre-se uma janela informano todos os argumentos possíveis dessa função. Isso torna muito dinâmico escrever linhas de comando, porque não precisaremos estar lembrando do nome dos argumentos de uma função, mas apenas entender o objetivo dessa função. Vejamos um exemplo a seguir.
Ambiente Global (ou área de trabalho, Workspace)
Quando usamos um comando de atribuição no console, o R armazena o nome associado ao objeto criado na área de trabalho (Workspace), que nós chamamos de Ambiente Global. Teremos uma seção introdutória na seção Ambientes e caminhos de busca, mas entendamos inicialmente que o objetivo de um ambiente é associar um conjunto de nomes a um conjunto de valores. Por exemplo:
# Nomes criados no ambiente
x <- 10 - 6; y <- 10 + 4; w <- "Maria Isabel"
# Verificando o nomes contidos no ambiente global
ls()
[1] "cran" "funcao" "github" "meu_nome"
[5] "n" "novo_ambiente" "rlink" "rstudio"
[9] "w" "x" "y" Observe que todos os objetos criados até o momento estão listados, e o que é mais surpreendente é que ambientes podem conter outros ambientes e até mesmo se conter. Observe o objeto meu_nome é um ambiente e está contido no Ambiente global. Será sempre dessa forma que recuperaremos um objeto criado no console do R. Caso contrário, se no console esse comando não for de atribuição esse objeto é perdido.
Arquivos .Rdata e .Rhistory
Ao final do que falamos até agora, todo o processo ao inserir linhas de comando do console, e desejarmos finalizar os trabalhos do ambiente R, dois arquivos são criados, sob a instrução do usuário em querer aceitar ou não, um .RData e outro .Rhistory, cujas finalidades são:
- .RData: salvar todos os objetos criados que estão atualmente disponíveis;
- .Rhistory: salvar todas as linhas de comandos inseridas no console.
Ao iniciar o R no mesmo diretório onde esses arquivos foram salvos, é carregado toda a sua área de trabalho anteriomente, bem como o histórico das linhas de comando utilizadas anteriormente.
Criando e salvando um script
A melhor forma de armazenarmos nossas linhas de código inseridas no console é criando um Script. Este é um arquivo de texto com a extensão “.R.” Uma vez criada, poderemos ao final salvar o arquivo e guardá-lo para utilizar futuramente.
No R, ao ser iniciado poderemos ir no menu em Arquivo > Novo script.... Posteriormente, pode ser inserido as linhas de comando, executadas no console pela tecla de atalho F5. As janelas do Script e console possivelmente ficarão sobrepostas. Para uma melhor utilização, estas janelas podem ficar lado a lado, configurando-as no menu em Janelas > Dividir na horizontal (ou Dividir lado a lado).
No RStudio, poderemos criar um Script no menu em File > New File > R Script, ou diretamente no ícone abaixo da opção File no menu, cujo o símbolo é um arquivo com o símbolo “+” em verde, que é o ícone do New File, e escolher R Script. Esse arquivo abrirá no primeiro quadrante na interface do RStudio.
Para salvar, devemos clicar no botão com o símbolo de disquete (R/RStudio), escolher o nome do arquivo e o diretório onde o arquivo será armazenado no seu computador. Algumas ressalvas devem ser feitas:
- Escolha sempre um nome sem caracteres especiais, com acentos, etc.;
- Escolha sempre um nome curto ou abreviado, que identifique a finalidade das linhas de comando escritas;
- Evite espaços se o nome do arquivo for composto. Para isso, use o símbolo underline “
_”; - Quando escrever um código, evite também escrever caracteres especiais, exceto em casos de necessidade, como imprimir um texto na tela, títulos na criação de gráficos, dentre outras. Nos referimos especificamente, nos comentários do código.
Um ponto bem interessante é o diretório. Quando criamos um Script a primeira vez, e trabalhamos nele a pós a criação, muitos erros podem ser encontrados de início. Um problema clássico é a importação de dados. O usuário tem um conjunto de de dados e deseja fazer a importação para o R, porém, mesmo com todos os comandos corretos, o console retorna um erro, informando que não existe esse arquivo que contém os dados para serem informados. Isso é devido ao diretório de trabalho atual. Para verificar qual o diretório que está trabalhando no momento, use a linha de comando:
getwd()
Para alterar o diretório de trabalho, o usuário deve usar a seguinte função setwd("Aqui, deve ser apontado para o local desejado!"). Supomos que salvamos o nosso Script tem sido em C:\meus_scripts_r. Assim, usamos a função setwd e ao apontarmos o local, as barras devem ser inseridas de modo invertido, isto é, setwd("C:/meu_scripts_r"), além de estar entre aspas.
No RStudio, isso pode ser feito em Session > Set Working Directory > To Source File Location. Isso levará ao diretório corrente do Script. Se desejar escolher outro diretório, vá em Session > Set Working Directory > Choose Directory.... Porém, uma vez criado um Script, e utilizado novamente, se o usuário estiver abrindo o RStudio também naquele primeiro momento, por padrão, o diretório de trabalho corrente será o mesmo do diretório do Script. Isso acaba otimizando o trabalho.
Devemos estar atentos também, quando trabalhamos utilizando Scripts ou arquivos de banco de dados, em locais diferentes do diretório corrente. Um outro recurso interessante é a função source(), que tem o objetivo de executar todas as linhas de comando de um Script sem precisar abri-lo. Isso pode ser útil, quando criamos funções para as nossas atividades, porém elas não se encontram no Script de trabalho para o momento. Assim, podemos criar um Script auxiliar que armazenas todas as funções criadas para as análises desejadas, e no Script corrente, poderemos chamá-las sem precisar abri o Script auxiliar. Todos os objetos passam a estar disponíveis no ambiente global.
Por fim, algo de muita importância para um programador, comente suas linhas de comando. Mas faça isso a partir do primeiro dia em que você criou o seu primeiro Script. Isso criará um hábito, uma vez que o arquivo não está sendo criado apenas para um momento, mas para futuras consultas. E quando voltamos a Scripts com muitas linhas de comando, principalmente depois de algum tempo, e sem comentários, possivelmente você passará alguns instantes para tentar entender o que foi escrito.
Outra coisa importante, é a boa prática de escrita de um código, e o RStudio nos proporciona algumas ferramentas interessantes. Mas isso será visto mais a frente.
Objetos
Definimos um objeto como uma entidade no ambiente R com características internas contendo informações necessárias para interpretar sua estrutura e conteúdo. Essas características são chamadas de atributos. Vamos entender o termo estrutura como a disposição de como está o seu conteúdo. Por exemplo, a estrutura de um objeto mais simples no R é um vetor atômico, pois os elementos contidos nele, apresenta o mesmo modo, um tipo de atributo. Falaremos nisso, mais à frente. De forma didática, adaptaremos a representação dos objetos no formato de diagrama.
Vejamos o seguinte código:
x <- 10
Todo mundo que tem uma certa noção sobre a linguagem R afirmaria: “criei um objeto x que recebe o valor 10.” Para Wickham (2019) essa afirmação é imprecisa e pode levar um entendimento equivocado sobre o que acontece de fato. Para o mesmo autor, o correto é afirmar que o objeto 10 está se ligando a um nome. E de fato, o objeto não tem um nome, mas o nome tem um objeto. O símbolo que associa um objeto a um nome é o de atribuição, <-, isto é, a junção do símbolo desigualdade menor e o símbolo de menos. Para ver qual objeto associado ao nome, o usuário precisa apenas digitar o nome no console e apertar a tecla ENTER.
Representaremos em termos de diagrama, um nome se ligando a um objeto, na Figura 8.
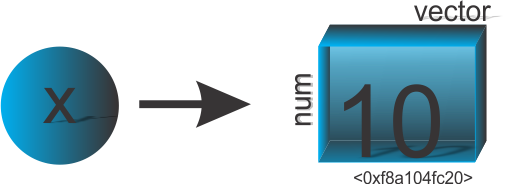
Figure 8: Dizemos que o nome 'x' se liga ao objeto do tipo (estrutura) vetor.
O identificador na memoría ativa desse objeto pode ser obtida por:
lobstr::obj_addr(x)
# [1] "0xf8a104fc20"
O diagrama explica que o nome criado “x” se associou com um objeto do tipo (estrutura) vetor (vector) e modo numérico (numeric)15, cuja identificação na memória ativa do seu computador foi <0xf8a104fc20>. É claro que para cada vez que o usuário abri o ambiente R e executar novamente esse comando, ou repeti o comando, esse identificador irá alterar.
Essa outra representação ficará mais claro para a afirmação feita anteriormente, no segundo diagrama, Figura 9, que representa a ligação do nome “y” ao mesmo objeto. Os termos nos diagramas, serão usados de acordo com a sintaxe da linguagem com os termos em inglês para melhor compreensão e fixação dos termos utilizados em R, uma vez que os termos na linguagem são baseados nesse idioma.
y <- x
lobstr::obj_addr(y)
# [1] "0xf8a104fc20"
Observem que não houve a criação de um outro objeto, mas apenas a ligação de mais um nome ao objeto existente, pois o identificador na memória ativa para o objeto não alterou, é o mesmo. Logo, não temos um outro objeto, mais dois nomes que se ligam ao mesmo objeto.
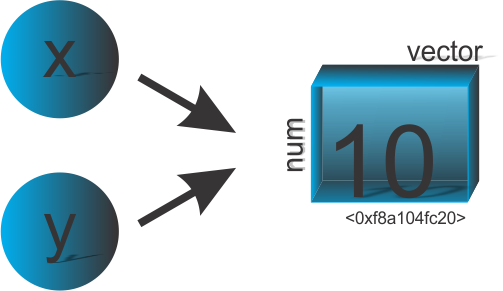
Figure 9: Dizemos que o nome 'x' e ‘y’ se ligam ao objeto do tipo (estrutura) vetor.
Mais especificamente, acrescentamos um outro diagrama, Figura 10, mostrando a representação do ambiente global (.GlobalEnv, nome associado ao objeto que representa o ambiente global).
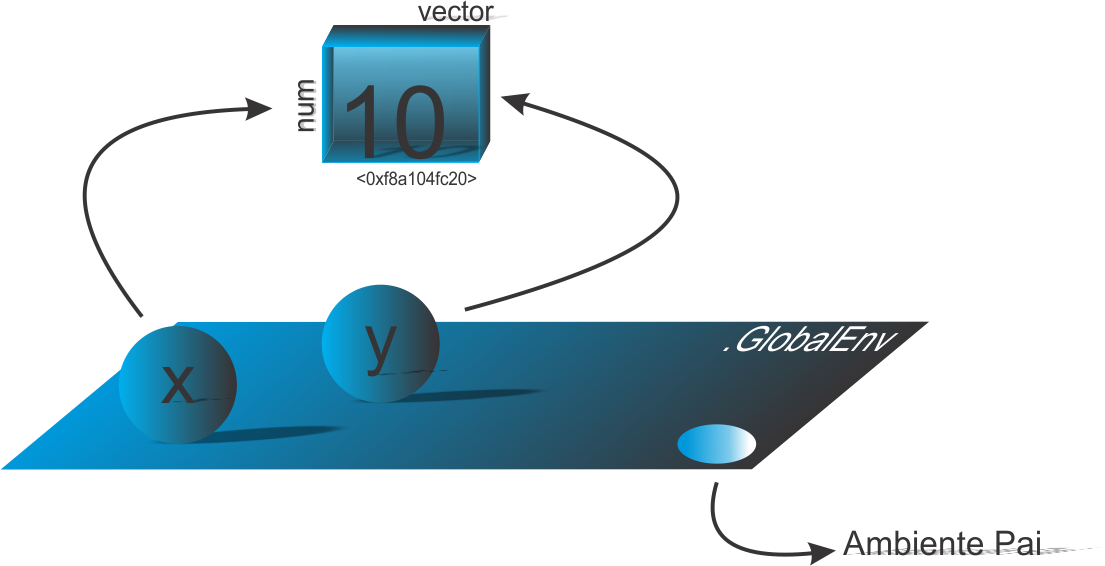
Figure 10: Dizemos que o nome 'x' e 'y' se ligam ao objeto do tipo (estrutura) vetor e essa ligação fica armazenada no ambiente global.
De todo modo, deixaremos para o módulo Programação em R (Nível Intermediário), uma abordagem mais profunda sobre o assunto.
O símbolo de atribuição poderá ser representado na direção da esquerda para à direita ou vice-versa, isto é,
x <- 10
10 -> x
Essas duas linhas de comando anteriores podem ter passado despercebidas pelo leitor em uma situação. Se na segunda linha tivéssemos alterado o valor do objeto de 10 para 30, por exemplo, a associação de x seria ao objeto 30. Isso significa que se o nome já existe, ele será apagado da memória ativa do computador e associado ao novo objeto. Veja,
Uma outra forma menos convencional é usar o comando assign, veja:
assign("m", 15)
m
[1] 15Ao invés do símbolo de atribuição, muitos usuários utilizam o símbolo da igualdade “=” para associarmos nomes aos objetos, que o ambiente R compreenderá. Contudo, discutiremos mas adiante, em Boas práticas de como escrever um código, que o uso da igualdade deverá em R ser usado apenas para a utilização em argumentos de uma função.
Quando desejamos executar mais de uma linha de comando por vez, separamos estas pelo símbolo “;” isto é,
x <- 10; w <- 15; x; w
[1] 10[1] 15Neste caso, executamos quatro comandos em uma linha. Associamos dois nomes a dois objetos e imprimimos os seus valores.
Por questão de comodidade, iremos a partir de agora, sempre nos referir a um objeto pelo nome associado a ele, para não está sempre se expressando como “um nome associado a um objeto.” Mas que fique claro a discussão realizada anteriormente sobre esses conceitos.
Nesse momento, nos limitaremos a falar sobre objetos que armazenam dados, do tipo caracteres, números e operadores lógicos (TRUE/FALSE).
Atributos
Todos os objetos, terão pelo menos dois tipos de atributos, chamados de atributos intrínsecos. Os demais atributos, quando existem, podem ser verificados pela função attributes(). A ideia dos atributos pode ser pensada como metadados, isto é, um conjunto de informações que caracterizam o objeto.
Diremos também que todos os objetos R tem uma classe, e por meio dessas classes, determinadas funções podem ter comportamento diferente a objetos com classes diferentes. Agora, devemos deixar claro essa informação, apesar do R seguir o princípio do Objeto, nem tudo é orientado a objetos, como por exemplo, observamos na linguagens C++ e Java. Deixemos esse tópico para o módulo Programação em R (Nível Intermediário).
A forma de se verificar a classe de um objeto é pela função class(). Contudo, os objetos internos do R (base), quando solicitado sua classe pela função class(), acabam retornando, algumas vezes, resultados equivocados. Uma alternativa é utilizar a função sloop::s3_class() do pacote sloop. Isso também será discutido no módulo Programação em R (Nível Intermedirário).
Devemos estar atentos a uma questão. Existe um atributo também chamado classe (“class”), e nem todos os objetos necessariamente tem esse atributo, apenas aqueles orientados a objetos, como é o caso do objeto com atributo classe . Por exemplo, é devido a classe factor no objeto criado pela função factor() que apesar do seu resultado ser numérico, este não se comporta como numérico. Isto significa que o atributo classe muda o comportamento de como funções veem esse objeto.
Para verificarmos se tal objeto tem o atributo class, usamos a função attributes(). Quando este atributo existe, ele é coincidente com o resultado obtido também pela função class().
O tipo das classes podem ser numeric, logical, character, list, matrix, array, factor e data.frame. Por enquanto, nos concentraremos nas classes de objetos que armazenam dados para uso em análises.
Para remover o efeito da classe, usamos a função unclass() para tal.
Por exemplo, quando criamos um objeto da classe data.frame, vejamos o seu comportamento:
# Criamos um objeto de classe 'data.frame'
dados <- data.frame(a = 1:3, b = LETTERS[1:3])
# Imprimindo na tela
dados
a b
1 1 A
2 2 B
3 3 C# Verificando sua classe
class(dados)
[1] "data.frame"# Verificando o efeito do objeto 'dados',
# sem o efeito da classe
dados2 <- unclass(dados); dados2
$a
[1] 1 2 3
$b
[1] "A" "B" "C"
attr(,"row.names")
[1] 1 2 3# Qual a classe desse objeto sem o efeito da
# classe 'data.frame'
class(dados2)
[1] "list"Observe que sem o atributo class= 'data.frame', o objeto tem classe list. Isto significa que, o objeto tem uma estrutura em forma de list, mas se comporta como um data.frame, que se apresenta como mostrado anteriormente.
Veremos no módulo Programação em R (Nível Intermediário) como criar atributos, classes, e mostrar que não conseguiremos mostrar todos os tipos de classes, pois a todo momento se cria classes em objetos R no desenvolvimento de pacotes.
Atributos intrínsecos
Todos os objetos tem dois atributos intrínsecos: o modo e comprimento. O modo representa a natureza dos elementos objetos. Para o caso dos vetores atômicos, o modo dos vetores podem ser cinco, numérico (numeric), lógico (logic), caractere16 (character), complexo (complex) ou bruto (raw). Este último, não daremos evidência para esse momento. O comprimento mede a quantidade de elementos no objeto.
Para determinarmos o modo de um objeto, usamos a função mode(). Vejamos:
# Objeto modo caractere
x <- "Ben"; mode(x)
[1] "character"# Objeto modo numerico
y <- 10L; mode(y)
[1] "numeric"# Objeto modo numerico
y2 <- 10; mode(y2)
[1] "numeric"# Objeto modo logico
z <- TRUE; mode(z)
[1] "logical"# Objeto modo complexo
w <- 1i; mode(w)
[1] "complex"Contudo, essa função mode() se baseou nos atributos baseados na linguagem S. Temos uma outra função para verificarmos o modo do objeto que é typeof(). O atributo modo retornado de um objeto para esta última função, está relacionado a tipagem de variáveis da linguagem C, uma vez que boa parte das rotinas no R está nessa linguagem, principalmente as funções do pacote base. Existem 25 tipos que serão detalhados no módulo Programação em R (Nível Intermedirário).
# Objeto modo caractere
x <- "Ben"; typeof(x)
[1] "character"# Objeto modo numerico (Inteiro)
y <- 10L; typeof(y)
[1] "integer"# Objeto modo numerico (Real)
y2 <- 10; typeof(y2)
[1] "double"# Objeto modo logico
z <- TRUE; typeof(z)
[1] "logical"# Objeto modo complexo
w <- 1i; typeof(w)
[1] "complex"Observamos que apesar de alguns vetores serem vazios, estes ainda tem um modo, observe nas seguintes linhas de comando:
# Vetor numérico vazio de comprimento 1
numeric(0)
numeric(0)[1] "numeric"[1] "double"# Vetor caractere vazio de comprimento 1
character(0)
character(0)[1] "character"[1] "character"A diferença existente nos objetos y e y2 para as funções mode() e typeof() se referem apenas como o R armazena essas informações na memória do computador. Podemos perguntar ao R se dois números são iguais, assim:
# 10 eh igual a 10L ?
10 == 10L
[1] TRUEVeja que o resultado é TRUE, isto é, sim eles são iguais. Agora, veja a próxima linha de comando:
# 10 eh identico a 10L ?
identical(10, 10L)
[1] FALSEO retorno agora foi FALSE, que significa que o armazenamento dessas informações não são iguais. Posteriormente, entenderemos no que isso reflete no código usuário, uma vez que um código escrito pode uma perda de desempenho simplesmente pela não necessidade de determinados objetos ou cópias realizadas.
O termo double retornado pela função typeof() significa dupla precisão na linguagem de programação, que acaba tenho uma exigência de mais memória do que o objeto de modo integer. Esses termos são utilizados na linguagem C. Já a linguagem S não os diferencia, utiliza tudo como numeric.
Aqui vale um destaque para o termo numérico, que no R podem ter três significados:
- Pode significar um número real, isto é, para a computação um número de dupla precisão (
numericedoubleseriam iguais nesse aspecto). Veja linha de código:
# Criacao de dois objetos de modo numerico
a <- numeric(1); b <- double(1)
# Verificando o modo
mode(a); mode(b)
[1] "numeric"[1] "numeric"# Verificando se 'a' e 'b' sao identificos
identical(a, b)
[1] TRUE- nos sistemas S3 e S4 (orientação a objetos), o termo numérico é usado como atalho para o modo
integeroudouble. Esse ponto veremos no módulo Programação emR(Nível Básico). Contudo, veja a linha de código:
- Pode ser utilizado (
is.numeric()) para verificar se determinados objetos tem o modo numérico. Por exemplo, temos um objeto de classefactorque é importante para a área da estatística experimental, representando os níveis de um fator em um experimento. Os elementos desse objeto pode ser número ou caracteres, mas serão representados como sempre por números. Entretanto, não se comportam como numérico. Veja a linha de comando:
# Criando um objeto de atributo classe 'factor':
fator <- factor("a"); fator
[1] a
Levels: a# O atributo classe muda a forma dos elementos. Veja quando retiramos o atributo
# classe 'factor', o objeto retorna o valor 1
unclass(fator)
[1] 1
attr(,"levels")
[1] "a"# Para confirmar essa afirmacao anterior, vejamos o modo
mode(fator)
[1] "numeric"typeof(fator)
[1] "integer"# Apesar do resultado retornar 1, veja que ele nao se comporta como numerico
is.numeric(fator)
[1] FALSEis.integer(fator)
[1] FALSEUma tabela a seguir, mostra o retorno dos seis principais modos de um objeto do tipo (estrutura) de vetores atômicos (Os modos apresentados baseiam-se apenas quanto a característica dos dados do objeto. É claro que um objeto não armazena apenas dados. Existem outras naturezas, que serão omitidas nesse momento):
typeof |
mode |
|---|---|
logical |
logical |
integer |
numeric |
double |
numeric |
complex |
complex |
character |
character |
raw |
raw |
O comprimento do objeto é informado pela função lenght(), do qual a representação em diagrama informa esse atributo. Vejamos as linhas de comando a seguir.
# Vetor de comprimento 5
v1 <- 1:5
# Vetor de comprimento 3
v2 <- c("Ben", "Maria", "Lana")
# Vetor de comprimento quatro
v3 <- c(TRUE, FALSE, TRUE, TRUE)
# Vejamos o comprimento dos vetores
length(v1)
[1] 5length(v2)
[1] 3length(v3)
[1] 4Um diagrama apresentando esses três objetos no ambiente global, pode ser apresentado na Figura 11. Observe que acrescentamos agora o comprimento dos objetos no diagrama entre colchetes, ao lado do atributo modo.

Figure 11: Objetos v1, v2 e v3.
Um resumo as funções mensionadas podem ser refletidas com as seguintes indagações:
base::class()eloop::c3_class(): Qual o tipo de objeto?base::mode(): Qual o tipo de dados baseados na linguagemS?base::typeof(): Qual o tipo de dados baseados na linguagemC?base::attributes(): O objeto tem atributos?base::length(): Qual o comprimento do objeto?
Usamos essa sintaxe pacote::nome_função() para entedermos qual o pacote da função que utilizamos. Contudo, essa forma tem uma importância no sentido de acesso a funções em um pacote sem necessitar anexá-lo no caminho de busca. Assunto abordado mais a frente.
Coersão
Como falamos anteriormente, os vetores atômicos armazenam um conjunto de elementos de mesmo modo. A coerção é a forma como o R coage o modo dos objetos. Por exemplo, se um elemento de modo caractere estiver em um vetor, todos os demais elementos serão convertidos para esse modo. Veja:
# Criando um objeto x e imprimindo o seu resultado
x <- c("Nome", 3, 4, 5);x
[1] "Nome" "3" "4" "5" Observa que todos os elementos ganharam aspas, isto é, se tornaram um caractere ou uma cadeia de caracteres. A coersão entre vetores de modo numeric, character e logical será sempre assim:

No caso dos vetores lógicos, todo TRUE se converterá em 1, e FALSE em 0. Porém, os modos dos vetores podem ser coagidos pelo usuário, usando as funções do tipo as.<modo ou tipo>() com prefixo as., isto é, se desejarmos que um objeto meu_objeto tenha o modo “character,” basta usar as.character(meu_objeto). Para desejar saber se um objeto é de um determinado modo, usamos as funções do tipo is.<modo ou tipo>(), com o prefixo is.. Vejamos,
# Objeto de modo numerico
minha_idade <- 35
mode(minha_idade)
[1] "numeric"# Coersão do objeto para modo caractere (`string`)
minha_idade <- as.character(minha_idade)
mode(minha_idade)
[1] "character"# Verificando se o objeto tem modo 'character'
is.character(minha_idade)
[1] TRUETipo de objetos
Por fim, pretendemos falar sobre os principais tipos de objetos. O tipo vamos entender como a estrutura de como os dados estão organizados em um objeto, relacionados aos seus atributos. Falamos anteriormente sobre a estrutura mais simples, que é o vetor atômico. Mas entendemos que um vetor em R podem ser considerados: atômicos ou listas. Podemos então subdividi-los em:
- Vetores atômicos:
- Lógicos, Numéricos e Caracteres;
- Matrizes unidimensionais (Matrix) e multidimensionais (Arrays);
- Vetores em listas:
- Listas (Lists);
- Quadro de dados (Data frames);
Existem outros, mas para esse módulo, exploraremos estes nas seções seguintes. As funções para as coersões realizadas pelos usuários, são similares as funções de coersão para modo, isto é, usar as funções prefixadas as.<tipo>.
Daremos uma visão geral dos objetos apresentados até o momento na Tabela a seguir.
| Objeto | Classe | Modo | São possíveis vários modos no mesmo objeto? |
|---|---|---|---|
| Vetor | numeric (integer ou double), character, complex, logical, raw |
numeric (integer ou double), character, complex, logical, raw |
Não |
| Matriz | matrix |
numeric (integer ou double), character, complex, logical, raw |
Não |
| Array | array |
numeric (integer ou double), character, complex, logical, raw |
Não |
| lista | list |
numeric (integer ou double), character, complex, logical, raw, expression, function |
Sim |
| Quadro de dados | data.frame |
numeric (integer ou double), character, complex, logical, raw |
Sim |
Vetores
Podemos dizer que existem três tipos principais de vetores atômicos:
- Numéricos (
numeric):- Inteiro (
integer); - Real (
double);
- Inteiro (
- Lógico (
logical); - Caractere (
character)
Existem dois tipos raros que são os complexos (complex) e brutos (raw), que falaremos no módulo seguinte.
Escalares
O menor comprimento de um vetor é de tamanho um, conhecido também como um escalar. Porém, para o R tudo é observado como um vetor. As sintaxes para os tipos especiais são: - os vetores lógicos assumem valores: TRUE ou FALSE, ou abreviados, T ou F, respectivamente. Existem valores especiais devido a precisão de operações na programação, que são os chamados pontos flutuantes. Nesse caso temos: Inf, -Inf e NaN, quando o resultados tende a \(\infty\), \(-\infty\), sem número, respectivamente;
# divisao de um numero por zero (+ infinito)
x <- 50 / 0; x
[1] Inf# divisao de um numero por zero (- infinito)
-50 / 0
[1] -Inf# Resultado sem número do tipo NaN
x - x
[1] NaN- os vetores numéricos do tipo
doublepodem ser representados de forma decima (0.123), científica (1.23e5), ou hexadecimal (3E0A); - os vetores numéricos do tipo
integersão representados pela letraLao final do número inteiro, isto é,1L,1.23e5L, etc.; - os caracteres são representados pelas palavras, letras, números ou caracteres especiais entre aspas, isto é,
"Ben","a". Pode ser utilizado também aspa simples,'Ben','a', etc.
Vetores longos
Os vetores longos podem ser criados pela função c() a incial da palavra concatenar, que significa agrupar. Vejamos alguns exemplos:
# Criando um vetor 'double'
vetor.num <- c(1, 2, 3, 4, 5); vetor.num
[1] 1 2 3 4 5typeof(vetor.num)
[1] "double"Uma coisa interessante é que por padrão, a função c() sempre cria um vetor de modo double, a menos que o usuário determine que estes elementos sejam inteiros, isto é,
[1] 1 2 3 4 5[1] "integer"Uma forma mais eficiente para criarmos um vetor com elementos de sequências regulares, é por meio da função primitiva (:), isto é, <menor valor da sequência>:<maior valor da sequência>. Vejamos:
# Criando uma sequência de 1 a 5
vetor.num3 <- 1:5; vetor.num3; typeof(vetor.num3)
[1] 1 2 3 4 5[1] "integer"Veremos mais a frente outras funções para construir sequências regulares. Se verificarmos os três objetos, veremos que todos eles são iguais:
vetor.num == vetor.num2
[1] TRUE TRUE TRUE TRUE TRUEvetor.num == vetor.num3
[1] TRUE TRUE TRUE TRUE TRUEvetor.num2 == vetor.num3
[1] TRUE TRUE TRUE TRUE TRUEO que vai difenrenciá-los é a forma de armazená-lo (double ou integer), e por consequência, o espaço na memória ativa. Veja:
# Objetos:
vetor.num <- c(1, 2, 3, 4, 5)
vetor.num2 <- c(1L, 2L, 3L, 4L, 5L)
vetor.num3 <- 1:5
# Memoria:
lobstr::obj_size(vetor.num)
96 Blobstr::obj_size(vetor.num2)
80 Blobstr::obj_size(vetor.num3)
680 BO que podemos observar é que o vetor de modo double precisa de mais memória do que o objeto de modo integer. O último objeto aparentemente ocupou mais memória. Contudo, essa função apresenta um recurso interessante apresentado nas versões posteriores R (3.5.0), que é chamado de abreviação alternativa. Esse recurso faz com que a sequência de números não seja armazenada completamente, apenas os extremos. Isso significa que para qualquer tamanho de sequência, a ocupação de memória do objeto será sempre a mesma. Outras formas de criar sequência de números é usando as funções rep() e seq(), do qual a ajuda pode ser realizada usando no console ?rep() e ?seq(), respectivamente.
Dessa forma, poderemos ter com o último objeto (vetor.num3) uma economia de memória, dependendo do tamanho do seu vetor, quando se compara com as outras opções. Veja:
# Tamanho de memoria dos objetos
lobstr::obj_size(1:10)
680 Blobstr::obj_size(1:10000)
680 Blobstr::obj_size(1:1000000)
680 B96 B40,048 B4,000,048 BManipulando vetores
Quando algum elemento de um vetor não está disponível, representamos pela constante lógica NA, que pode ser coagida para qualquer outro modo de vetor, exceto para raw. Podemos ter constantes lógicas NA específicas para modos específicos: NA_integer_, NA_real_ (o equivalente para o modo double), NA_complex_ e NA_character_. Entretando, dependendo de onde o NA é inserido, o atributo modo no objeto já converte para NA específico de acordo com o seu atributo modo. Essa constante contido no vetor não altera o modo do vetor, isto é,
[1] "double"[1] "double"[1] "character"Podemos criar vetores atômicos iniciais sem nenhuma elemento, por meio das funções numeric(0), character(0) e logical(0), isto é,
[1] 0[1] 0[1] 0Para inserirmos valores a esses vetores usamos o sistema de indexação, que no caso da linguagem R, o contador começa a partir do número 117. Vejamos,
[1] 0[1] 0[1] 0# Vetor numerico de comprimento 0
v1 <- numeric(0)
v2 <- character(0)
v3 <- logical(0)
# Inserimos 3 elementos em v1 e depois imprimos o seu resultado
v1[1] <- 5; v1[2] <- 3; v1[3] <- 10; v1; length(v1)
[1] 5 3 10[1] 3Assim, como exercício vocês podem completar para os dois outros vetores. Uma vez criado o vetor, se desejarmos acessar os seus elementos, usamos também o sistema de indexação:
# Vetor numerico de comprimento 0
v1 <- numeric(0)
v2 <- character(0)
v3 <- logical(0)
# Inserimos 3 elementos em v1 e depois imprimos o seu resultado
v1[1] <- 5; v1[2] <- 3; v1[3] <- 10
# Imprimindo apenas o primeiro valor
v1[1]
[1] 5# Imprimindo os dois ultimos
v1[2:3]; v1[c(2, 3)]
[1] 3 10[1] 3 10# Imprimindo todos
v1
[1] 5 3 10Aritmética e outras operações
As operações com vetores não necessariamente são as operações realizadas baseadas na álgebra de matrizes. O que a linguagem R faz é realizar as operações elemento a elemento, mantendo o comprimento de tamanho igual ao tamanho do maior vetor na operação. Vejamos as operações aritméticas entre vetores de tamanho 1:
# Soma de dois vetores
2 + 3
[1] 5# Exceto pela sintaxe, '+' eh uma chamada de funcao
`+`(2, 3)
[1] 5# Subtracao de dois vetores
3 - 2
[1] 1# Exceto pela sintaxe, '-' eh uma chamada de funcao
`-`(3, 2)
[1] 1# Multiplicacao de dois vetores
3 * 2
[1] 6# Exceto pela sintaxe, '*' eh uma chamada de funcao
`*`(3, 2)
[1] 6# Divisao de dois vetores
3 / 2
[1] 1.5# Exceto pela sintaxe, '/' eh uma chamada de funcao
`/`(3, 2)
[1] 1.5Essas mesmas operações podem ser realizadas elemento a elemento para vetores de comprimento maior que 1, observe:
[1] 5 7 9[1] 5 7 9[1] 4 10 18[1] 4.0 2.5 2.0Quando os vetores não têm mesmo comprimento, o R completará de forma sequencial o menor vetor até que ele atinja o tamanho do maior vetor, observe:
# Soma de vetores de comprimento diferente
1:10 + 3:10
[1] 4 6 8 10 12 14 16 18 12 14O segundo vetor repetiu os elementos 3, 4, 5, isto é, os três primeiros elementos do vetor, para que o seu comprimento se tornasse igual ao comprimento do primeiro vetor. Após isso, foi realizado a soma elemento a elemento. Esse procedimento ocorre com os demais tipos de operações.
Demais operações podem ser realizadas de acordo com as funções apresentadas na Tabela abaixo.
| Função (Ou operador) | Finalidade |
|---|---|
+ |
Soma unária, por exemplo (+ 4), ou binária entre dois vetores |
- |
Subtração unária, por exemplo (- 3), ou binária entre dois vetores |
* |
Multiplicação entre dois vetores |
/ |
Divisão entre dois vetores |
^ ou ** |
Expoenciação binária, isto é 2^3 ou 2 ** 3 |
%/% |
Divisão inteira |
%% |
Restante da divisão |
sum() |
Soma de elementos de um vetor |
prod() |
Produtório dos elementos de um vetor |
sqrt() |
Raiz quadrada dos elementos de um vetor |
log() |
Função Logaritmo neperiano |
log10() |
Função Logaritmo na base 10 |
exp() |
Função exponencial |
mean() |
Média dos elementos de um vetor |
sd() |
Desvio padrão dos elementos de um vetor |
var() |
Variância dos elementos de um vetor |
median() |
Mediana dos elementos de um vetor |
round() |
Arredondamento de vetor numérico. Outros tipos são: trunc(), floor() e ceiling(). |
Demais funções podem ser procuradas no manual An Introduction to R, ou execute no console ?Arithmetic.
Operadores lógicos
Os operadores lógicos têm a função de avaliar determinada condição e retornar TRUE/FALSE. São eles:
| Operador Lógico | Sintaxe | Pergunta |
|---|---|---|
< |
a < b |
a é menor que b? |
> |
a > b |
a é maior que b? |
== |
a == b |
a é igual b? |
!= |
a != b |
a é diferente b? |
>= |
a >= b |
a é maior ou igual a b? |
<= |
a <= b |
a é menor ou igual a b? |
%in% |
"a" %in% c("a", "b", "c") |
O elemento "a" está no vetor c("a", "b", "c")? |
A operação binária significa que a função exige dois argumentos (ou operandos), isto é, <Argumento 1> <Operador> <Argumento 2>. Para mais detalhes, use no console ?Syntax.
Vejamos alguns exemplos:
# Operador '>' entre vetores de comprimento 1
1 > 3
[1] FALSE# Operador '<' com vetor de comprimento maior que 1
1 < c(0, 1, 3)
[1] FALSE FALSE TRUE[1] FALSE TRUE FALSE# Operador '%in%' verificando se os elementos do primeiro vetor
# estao no segundo vetor
1 %in% c(3, 4, 5)
[1] FALSE# Operador '%in%' verificando se os elementos do primeiro vetor
# estao no segundo vetor
c(1, 2) %in% c(3, 4, 5)
[1] FALSE FALSE# Operador '%in%' verificando se os elementos do primeiro vetor
# estao no segundo vetor
c(1, 2, 3) %in% c(3, 4, 5)
[1] FALSE FALSE TRUE# Operador '%in%' verificando se os elementos do primeiro vetor
# estao no segundo vetor
c(1, 2, 3, 4) %in% c(3, 4, 5)
[1] FALSE FALSE TRUE TRUEO que é interessante nesse operador %in%, que na realidade é uma função com dois argumentos, constitui uma forma de criar operadores binários especiais do tipo %<nome_sintatico>%, que esse tipo de função é uma das mais conhecidas hoje na análise de dados usando o operador pipe (%>%) do pacote magrittr da família de pacotes Tidyverse. A diferença no operador pipe é que o segundo operando (Argumento 2) é uma função que recebe no primeiro argumento o operando 1 (Argumento 1). Por fim, o operador %>% acaba sendo um operado unário. Veremos mais detalhes na seção sobre criação de funções.
Operadores Booleanos
O operadores booleanos avaliam diversas operações lógicas (condições) para ao final retornar um TRUE/FALSE. Vejamos a Tabela a seguir, com esses operadores e suas indagações.
| Operador Booleano (ou Função) | Sintaxe | Pergunta |
|---|---|---|
& ou && |
cond1 & cond2 |
As cond1 e cond2 são verdadeiras? |
| ou || |
cond1 | cond2 |
A cond1 ou cond2 é verdadeira? |
xor() |
xor(cond1, cond2) |
Apenas a cond1 ou a cond2 é verdadeira? |
! |
!cond1 |
É falso a cond1 ? |
any() |
any(cond1, cond2, ...) |
Alguma das condições são verdadeiras? |
all() |
all(cond1, cond2, ...) |
Todas as condições são verdadeiras? |
Vejamos alguns exemplos:
# Criando objetos
x <- 1:3
y <- 1:3
z <- c(1, 2, 4)
# Primeira condicao
x == y
[1] TRUE TRUE TRUE# Segunda condicao
y == z
[1] TRUE TRUE FALSE# Terceira condicao
x == y & y == z
[1] TRUE TRUE FALSEFica como sugestão de exercício os leitores criarem condições para os demais operadores booleanos.
Matrizes bidimensionais
A apresentação dos próximos objetos daqui pra frente, desde matrizes até quadro de dados (data frame) não é apresentar todas as manipulações possíveis sobre esses objetos. Mas mostrar a sua estrutura e condições básicas imposta sobre eles. Assim, não apresentaremos funções para manipulações com matrizes, por exemplo, porque isso não é o propósito do curso. Daremos a ideia de uma matriz é na realidade um vetor bidimensional, assim como um quadro de dados que na realidade é uma lista.
Quando usamos um atributo chamado dim em um vetor atômico, criamos na realidade vetores bi ou multidimencionais, isto é, objetos do tipo matrizes ou arrays. Observe o que falados anteriormente, o atributo pode mudar a estrutura do objeto.
Vejamos alguns exemplos:
# Criando um vetor atomico
x <- 1:6; x
[1] 1 2 3 4 5 6# Verificando se o objeto 'x' tem atributo adicionado
attributes(x)
NULL# Vamos verificar a classe do objeto x
sloop::s3_class(x)
[1] "integer" "numeric"# Adicionando o atributo dim
dim(x) <- c(2, 3) # 2 x 3 = 6 (Comp do vetor)
# attr(x, "dim") <- c(2, 3)
# Observando agora o comportamento do objeto 'x'
x
[,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6# Verificando novamente se 'x' tem atributo
attributes(x)
$dim
[1] 2 3# Verificando a classe do objeto
sloop::s3_class(x)
[1] "matrix" "integer" "numeric"O atributo dim recebeu uma informação bidimensional, isto é, o número de linhas e colunas, respectivamente. Uma outra forma para construir uma matriz é usando a função matrix, que de modo similar, temos:
# Criando uma matriz
matrix(1:6, 2, 3)
[,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6# Criando uma matriz
matrix(1:6, 2, 3, byrow = TRUE)
[,1] [,2] [,3]
[1,] 1 2 3
[2,] 4 5 6Se desejássemos que os números fossem inserido em linhas e não iniciados pelas colunas, usaríamos o argumento byrow = TRUE, isto é,
# Criando uma matriz
matrix(1:6, 2, 3, byrow = TRUE)
[,1] [,2] [,3]
[1,] 1 2 3
[2,] 4 5 6Para acessarmos ou alterarmos os elementos de uma matriz, usamos o sistema de indexação similar ao vetor, porém, devemos indexar as linhas e colunas. Por exemplo, o elemento da primeira linha e primeira coluna pode ser obtido por x[1, 1], e assim por diante. Todos os elementos da linha 1, x[1,], ou todos os elementos da coluna 1, x[,1].
Matriz multidimensional
A ideia do objeto matriz multidimencional (ou array) é similar ao da matriz, a diferença é que agora é um vetor atômico de mais de duas dimensões. Vejamos,
# Criando um vetor atomico
x <- 1:12; x
[1] 1 2 3 4 5 6 7 8 9 10 11 12# Verificando se o objeto 'x' tem atributo adicionado
attributes(x)
NULL# Vamos verificar a classe do objeto x
sloop::s3_class(x)
[1] "integer" "numeric"# Adicionando o atributo dim
dim(x) <- c(2, 3, 2) # 2 x 3 x 2 = 12 (Comp do vetor x)
# attr(x, "dim") <- c(2, 3, 2)
# Observando agora o comportamento do objeto 'x'
x
, , 1
[,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6
, , 2
[,1] [,2] [,3]
[1,] 7 9 11
[2,] 8 10 12# Verificando novamente se 'x' tem atributo
attributes(x)
$dim
[1] 2 3 2# Verificando a classe do objeto
sloop::s3_class(x)
[1] "array" "integer" "numeric"Observe que criamos duas matrizes de dimensão (2 x 3). Para acessar os elementos desse objeto, usaremos também o sistema de indexação, agora acrescentando a terceira dimensão. Por exemplo, para acessar o elemento da linha 1, coluna 1, matriz 1, temos x[1, 1, 1], ou todos os elementos da linha 1, matriz 1, temos x[1, , 1].
Uma outra forma de criar um objeto array é usar a função array(). De modo similar, temos:
, , 1
[,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6
, , 2
[,1] [,2] [,3]
[1,] 7 9 11
[2,] 8 10 12Um exercício para os leitores seria, como poderíamos criar um array de 4 dimensões?
Mostramos um quadro resumo de funções que podem ser utilizadas (Wickham (2019)). Para auxílio nas funções, use sempre o símbolo de interrogação antes das funções e execute no console. Por exemplo, ajuda da função names(), use ?names().
| Vetor | Matriz | Array |
|---|---|---|
names() |
rownames(), colnames() |
dimnames() |
length() |
nrow(), ncol() |
dim() |
c() |
rbind(), cbind() |
abind::abind() |
- |
t() |
aperm() |
is.null(dim(x)) |
is.matrix() |
is.array |
Listas de dados
As listas são como vetores atômicos, porém mais complexos, isto é, os elementos de uma lista são vetores atômicos, como também outras listas, funções, expressões. Esta última é o que chamamos de objetos recursivos. A forma de se obter uma lista é pela função list(). Vejamos os comandos a seguir.
# Criando uma lista
l0 <- list(1:3, letters[5], list(1, 2, 3),
mean, expression(x ~ y))
# Imprimindo a lista
l0
[[1]]
[1] 1 2 3
[[2]]
[1] "e"
[[3]]
[[3]][[1]]
[1] 1
[[3]][[2]]
[1] 2
[[3]][[3]]
[1] 3
[[4]]
function (x, ...)
UseMethod("mean")
<bytecode: 0x0000000008e55c60>
<environment: namespace:base>
[[5]]
expression(x ~ y)Podemos acessar ou alterar os elementos de uma lista por meio do operador $, ou pelo sistema de indexação, que diferencia um pouco da indexação dos vetores. Por exemplo, o primeiro elemento desse vetor pode ser acessado por l0[[1]], o terceiro l0[[3]], e assim por diante. Para acessar informações específicas dentro dos elementos, usamos l0[[3]][2], isto é, imprimimos o segundo valor do segundo elemento. Os elementos de um lista são na realidade outros objetos, do qual conseguimos acessar também os elementos desses objetos.
Quando nominamos os objetos contidos nas listas, podemos utilizar o operador $, para acessar esses objetos. Vejamos,
# Criando uma lista
l0 <- list(l01 = 1:3,
l02 = letters[5],
l03 = list(1, 2, 3),
l04 = mean,
l05 = expression(x ~ y))
# Imprimindo o primeiro elemento (objeto) da lista 'l0'
l0$l01
[1] 1 2 3# Imprimindo o segundo
l0$l02
[1] "e"As listas têm importâncias diversas dentro da linhagem R, por exemplo, o atributo em um objeto é armazenado em forma de lista. A coerção sempre força um vetor atômico a uma lista. Vejamos as linhas de comando a seguir.
# Vejamos as linhas de comando
l1 <- list(list(1, 2), c(3, 4))
l2 <- c(list(1, 2), c(3, 4))
# Vejamos as suas estruturas
str(l1)
List of 2
$ :List of 2
..$ : num 1
..$ : num 2
$ : num [1:2] 3 4str(l2)
List of 4
$ : num 1
$ : num 2
$ : num 3
$ : num 4Observamos no objeto l1, temos uma lista cujos elementos são outra lista, o elemento 3 e o elemento 4. O vetor c(3, 4) se transformou em dois elementos de l1. No objeto l2, poderíamos pensar que como a lista está dentro da função c(), os elementos da lista fariam parte dos elementos de um vetor. Porém isso não ocorre. O que temos é uma coerção em que a lista força ao vetor a se tornar lista. Por fim, temos em l2 quatro elementos em uma lista.
Quadro de dados
O objeto quadro de dados (Data frame) é uma lista com classe data.frame, em que contém dois atributos. Porém, com algumas restrições:
- Os componentes devem ser vetores uni ou multidimencionais, listas ou até mesmo quadro de dados;
- As colunas das matrizes, listas ou quadro de dados são inseridas como colunas do quadro de dados;
- A partir da versão R (4.0.0), os vetores terão mesmo modo no quadro de dados. Antes os vetores em modo caractere eram convertidos em objeto do tipo fator. Para convertê-lo automaticamente use o argumento
stringsAsFactors = TRUE. Por sugestão, prefira a mudança usando a funçãofactor(), para ter um maior controle dos níveis; - Os objetos inseridos no quadro de dados devem ter o mesmo comprimento.
Para criarmos um objeto do tipo quadro de dados (data frame), usamos a função data.frame(). Assim, como nas listas podemos inserir os objetos no quadro de dados inserindo o nome nas colunas ou não. A forma de acessar os elementos é interessante, podemos usar a sintaxe de indexação de uma lista ou de uma matriz. Vejamos os exemplos a seguir.
# Criando um quadro de dados
dados <- data.frame(x = 1:10,
y = letters[1:10],
z = rep(c(TRUE, FALSE), 5))
# Imprimindo dados
dados
x y z
1 1 a TRUE
2 2 b FALSE
3 3 c TRUE
4 4 d FALSE
5 5 e TRUE
6 6 f FALSE
7 7 g TRUE
8 8 h FALSE
9 9 i TRUE
10 10 j FALSE# Acessando os elementos de forma de lista
dados[[1]]
[1] 1 2 3 4 5 6 7 8 9 10dados$x
[1] 1 2 3 4 5 6 7 8 9 10# Acessando os elementos em forma de matriz
dados[1, ] # Coluna 1
x y z
1 1 a TRUEdados[1, 1] # Elemento da linha 1 coluna 1
[1] 1dados[, 1] # Linha 1
[1] 1 2 3 4 5 6 7 8 9 10Quando importamos um conjunto de dados, por exemplo usando a função read.table(), o objeto que armazena esses dados é um quadro de dados. Assunto discutido mais a frente.
A semelhança com a forma retangular de uma matriz, faz com que algumas funções utilizadas em matrizes sejam utilizadas em quadro de dados:
- As funções
rownames()ecolnames(), retornam ou inserem os nomes das linhas e colunas, respectivamente. A funçãonames()retorna o nome das colunas. - A dimensão das linhas e colunas podem ser obtidas pelas funções
nrow()encol(), respectivamente. A funçãolength()retorna o número de colunas.
Em algumas situações, estamos interessados em otimizar o nosso tempo de programação, e achamos muito demorado ou não conveniente a utilização da sintaxe objeto$elemento para acessar os elementos de uma lista. Dessa forma, poderemos utilizar a função attach() para que os elementos do quadro de dados estejam disponíveis (anexados) no caminho de busca, e assim, possamos acessar os elementos (ou objetos) do quadro de dados sem precisar mencioná-lo. Vejamos,
# Criando um quadro de dados
dados <- data.frame(x = 1:10,
y = letters[1:10],
z = rep(c(TRUE, FALSE), 5))
# Usando a funcao attach()
attach(dados)
# Acessando os elementos
x; y; z
, , 1
[,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6
, , 2
[,1] [,2] [,3]
[1,] 7 9 11
[2,] 8 10 12[1] 1 2 3[1] 1 2 4# Desanexando dados
detach(dados)
Essa função attach() tem implicações, quando por exemplo se deseja inseri-la na construção de um pacote R. Iremos discutir esse ponto mais a frente. Para desanexar o quadro de dados, use detach(). A função attach() é genérica e pode ser usada em qualquer objeto de modo list ser anexado no caminho de busca.
Importando dados
A importação/exportação de dados era algo que em poucas linhas conseguíamos explicar sobre o ambiente R, no sentido de análise de dados. Entretanto, observando o terceiro princípio do R, afirmado por Chambers (2016):
- Princípio da Interface: Interfaces para outros programas são parte do
R
Hoje é uma realidade a interação que o ambiente R tem com outras interfaces (programas, linguagens, etc.). A facilidade em utilizar outras linguagens dentro do ambiente R torna assim mais complexo a importação/exportação de dados, uma vez que o objetivo do R, apesar do R Core Team ainda limitar a sua definição como o ambiente para a computação estatística, a ferramenta se tornou tão versátil, que hoje torna humilde essa definição. Para mais detalhes acesse o manual R Data Import/Export. Um outro fator e tema atual é a era dos grandes bancos de dados (Big Data), do qual se tem um grande conjunto de variáveis e necessitamos fazer a importação por APIs, por exemplo, ou outras vias. Temas como esses, abordaremos no módulo Programação em R (Nível Avançado).
Nesse momento, limitaremos esse assunto ao objetivo de termos um conjunto de dados em arquivos de texto (extensões do tipo .txt, .csv, xls), formato binário (.xls ou .xlsx) ou digitados manualmente pelo teclado do computador. Assim, a primeira forma de como os dados estão dispostos, precisaremos importá-los e armazená-los em um quadro de dados (data frame), para que esteja disponível na área de trabalho (ambiente global) do R, e dessa forma, possamos utilizá-lo. Ao final do tratamento dos dados, podemos exportar essas informações para arquivos externos, e daí também, usaremos os arquivos de textos e o formato binário (.xls), mensionados anteriormente.
Preparação dos dados
A primeira coisa que devemos entender quando desejamos construir o arquivo de dados, é entender que sempre organizaremos as variáveis em colunas, com os seus valores em linhas, Figura 12. Sempre a primeira linha das colunas representarar o nome das variáveis. Esse é outro ponto importante, pois devemos ter a noção que alguma linguagem irá ler esse banco de dados. Assim, quanto mais caracteres diferentes do padrão ASCII, mais difícil será a leitura desses dados. Assim, sugerimos alguns padrões:
- Evitem símbolos fora do padrão alfanumérico;
- Evitem mistura de letras minúsculas com letras maiúsculas. Isso facilitará o acesso a essas variáveis. Contudo, lembre-se do padrão de nomes sintéticos permissíveis do R;
- Lembre-se que o banco de dados será utilizado para que um programa faça a sua leitura, portanto, deixe a formatação da apresentação dos dados para arquivos específico. Sendo assim, evitem comentários nesses arquivos, ou qualquer outro tipo de informação que não seja o banco de dados;
- Evitem palavras longas, por exemplo,
segundavariavel(má escolha),segvar(boa escolha),seg_var(boa escolha); - Evitem palavras compostas com espaço entre elas. Para isso use o símbolo
_, por exemplo,var 2(má escolha),var2(boa escolha),var_2(boa escolha);
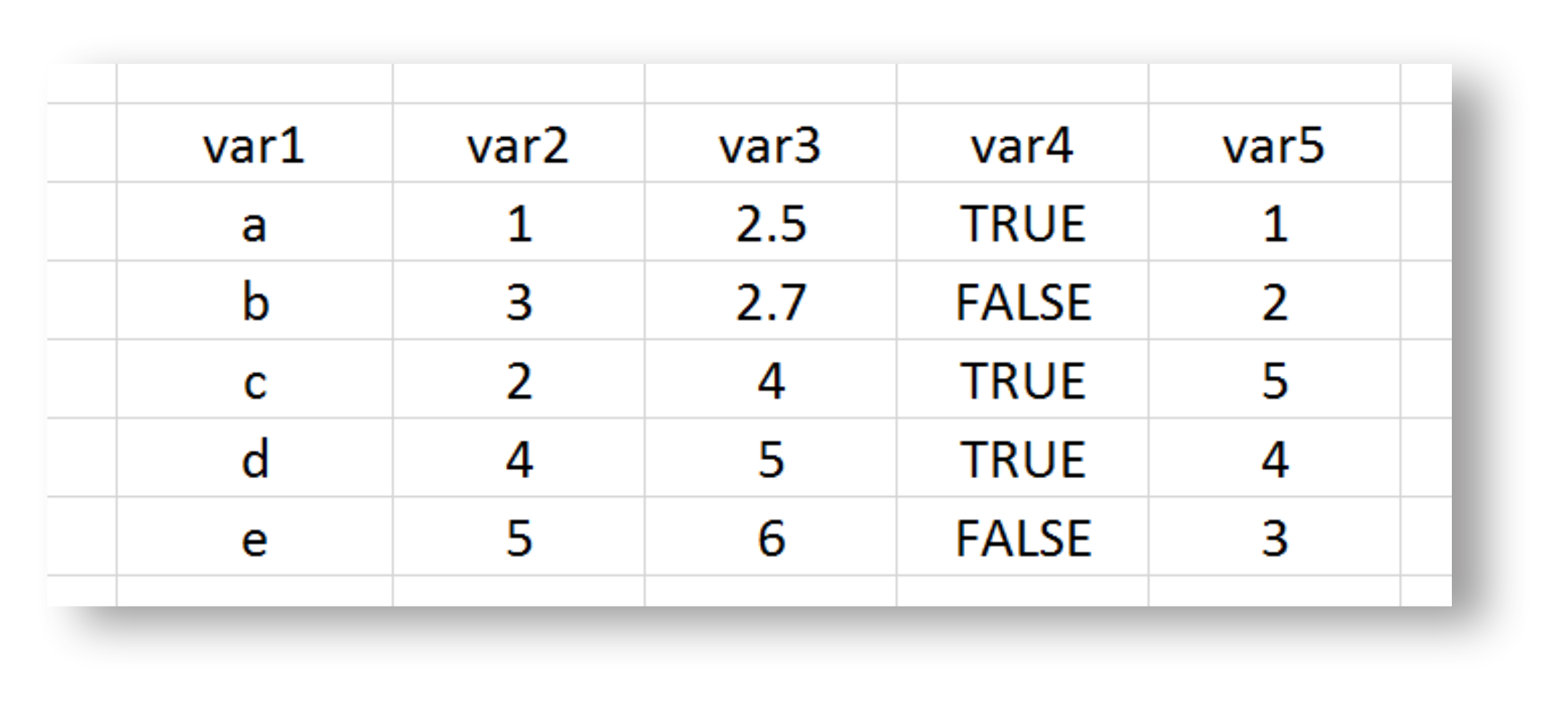
Figure 12: Modelo estrutural de um banco de dados.
Importando dados
A função primária responável pela importação de dados é a função scan(). Por exemplo, funções como read.table(), read.csv() e read.delim(), usam a função scan() em seu algoritmo.
A primeira ideia sobre importação de dados pode ser inserindo-os pelo teclado no próprio ambiente R. Para isso, usaremos a função scan(). Vejamos,
# Criando e inserido os elementos do objeto dados
x <- scan()
Após executado essa linha de comando, aparecerá no console 1: que significa, digitar o primeiro valor do objeto x, e depois clicar em ENTER. Depois 2:, que significa digitar o segundo valor, e clicar em ENTER. Depois de inserido todos os valores necessários, aperte a tecla ENTER duas vezes no console, para sair da função scan().
O mais tradicional é usar programa para criação de banco de dados e deixá-lo pronto para o R lê-lo. O tipo de arquivo de texto que melhor controla a separação de variáveis é com a extensão .csv, uma vez que separamos as variáveis por “;” é o padrão. O arquivo de texto com extensão .txt, geralmente usa espaços. Isso acaba gerando problema de leitura no R, porque muitos usuários usam nomes de variáveis muito grandes, palavras compostas, de forma a desalinhar as colunas das variáveis. Daí, como a separação das variáveis é por meio de espaços, acaba gerando problema de leitura. Uma outra forma, é fazer importação de dados gerados pelo próprio R, extensão .RData.
Temos a opção de usar um editor de banco de dados para essas extensões por meio de programas como MS Excel, Libre Office, dentre outros. Estes exportam arquivos binários do tipo .xls, .xlsx, dentre outros. Uma sugestão para diminuir complicações, é exportar os bancos de dados para arquivos de texto sitados acima, que também é possível ser exportado por esses programas. Isso evita a necessidade de ser instalado mais pacotes e dor de cabeça. Porém, para quem ainda deseja enfrentar, sugerimos a leitura do pacote readr, como exemplo, porém existem diversos outros pacotes para este mesmo fim.
Uma vez que o banco de dados está pronto, a leitura destes pode ser feita por alguns caminhos. Mostraremos o mais trivial que é o botão Import Dataset, terceiro quadrante, aba Environment, na IDE do RStudio. Veja na Figura 13.
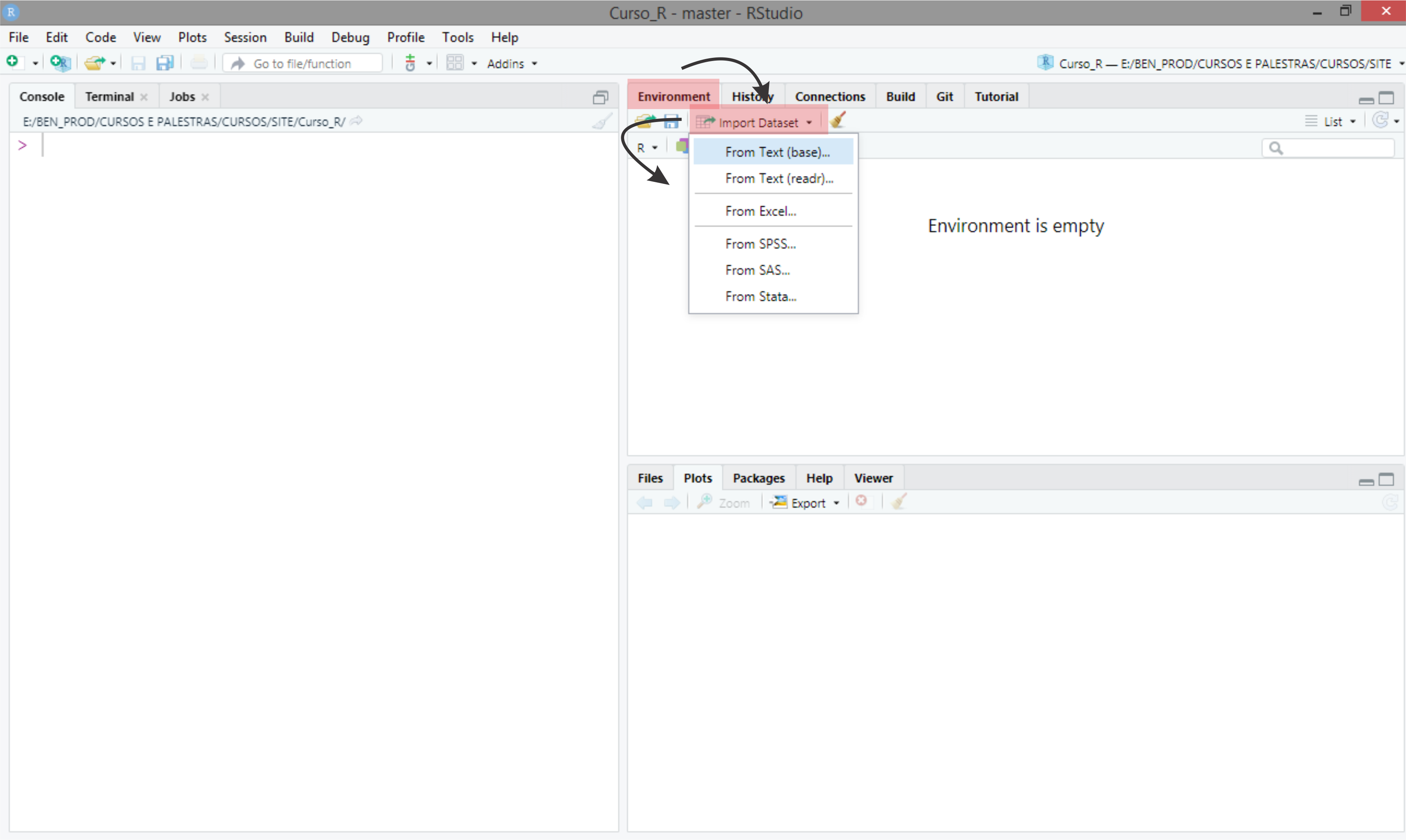
Figure 13: Usando o RStudio para importar dados.
Posteriormente, indique o arquivo para leitura. Aparece algumas opções de tipo de arquivo. Em nosso caso, usaremos a opção From Text (base), que significa realizar a leitura para os tipos de arquivo .txt ou .csv. Daí os passos seguintes são:
- Escolher o arquivo para leitura dos dados;
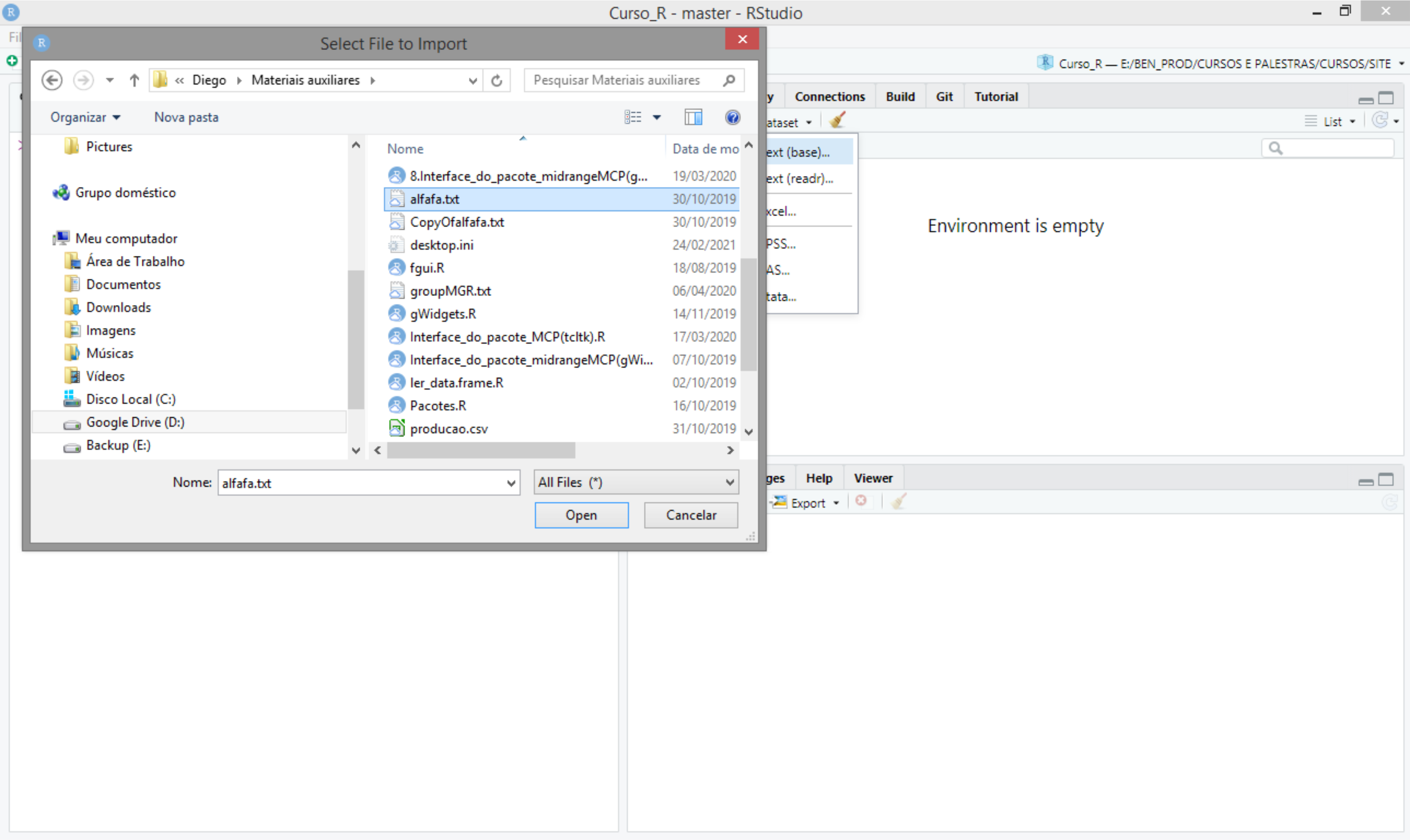
Figure 14: Usando o RStudio para importar dados.
- Configurar a leitura do banco de dados. Uma prévia pode ser vista no quadro Data Frame. Se for visualizado, algum problema, isso significa que deve ser informado opções adicionais como separador de variáveis (Separator), símbolo para casas decimais (Decimal), dentre outras opções. Por fim, digitar o nome associado ao objeto (Name) que será criado do tipo quadro de dados (data frame), e clicar no botão Import;
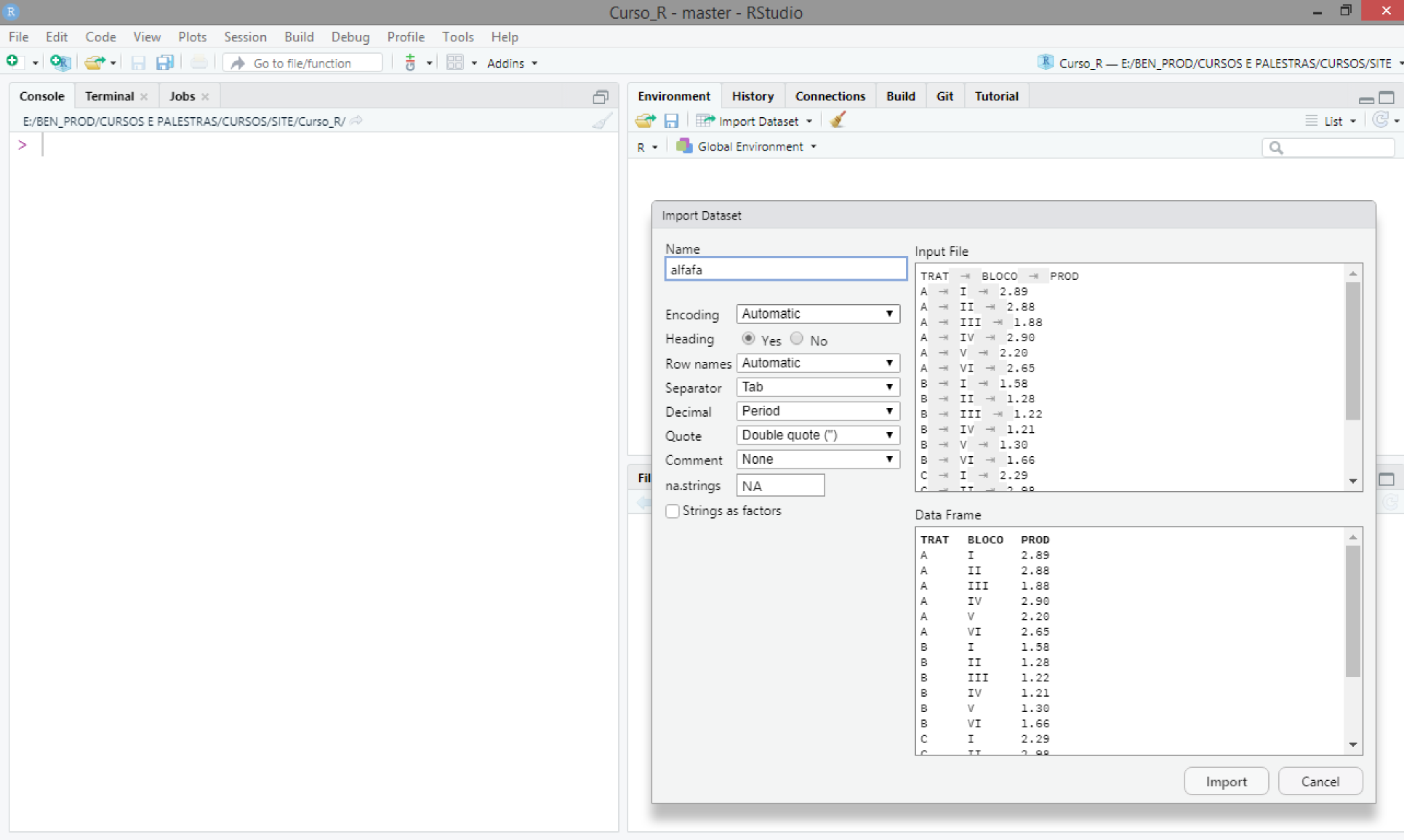
Figure 15: Usando o RStudio para importar dados.
- Uma vez inserido, o RStudio apresenta a linha de comando utilizada para importar os dados no console (2º quadrante), o conjunto de dados (1º quadrante), e a ligação entre o nome e o objeto no ambiente global (3º quadrante).
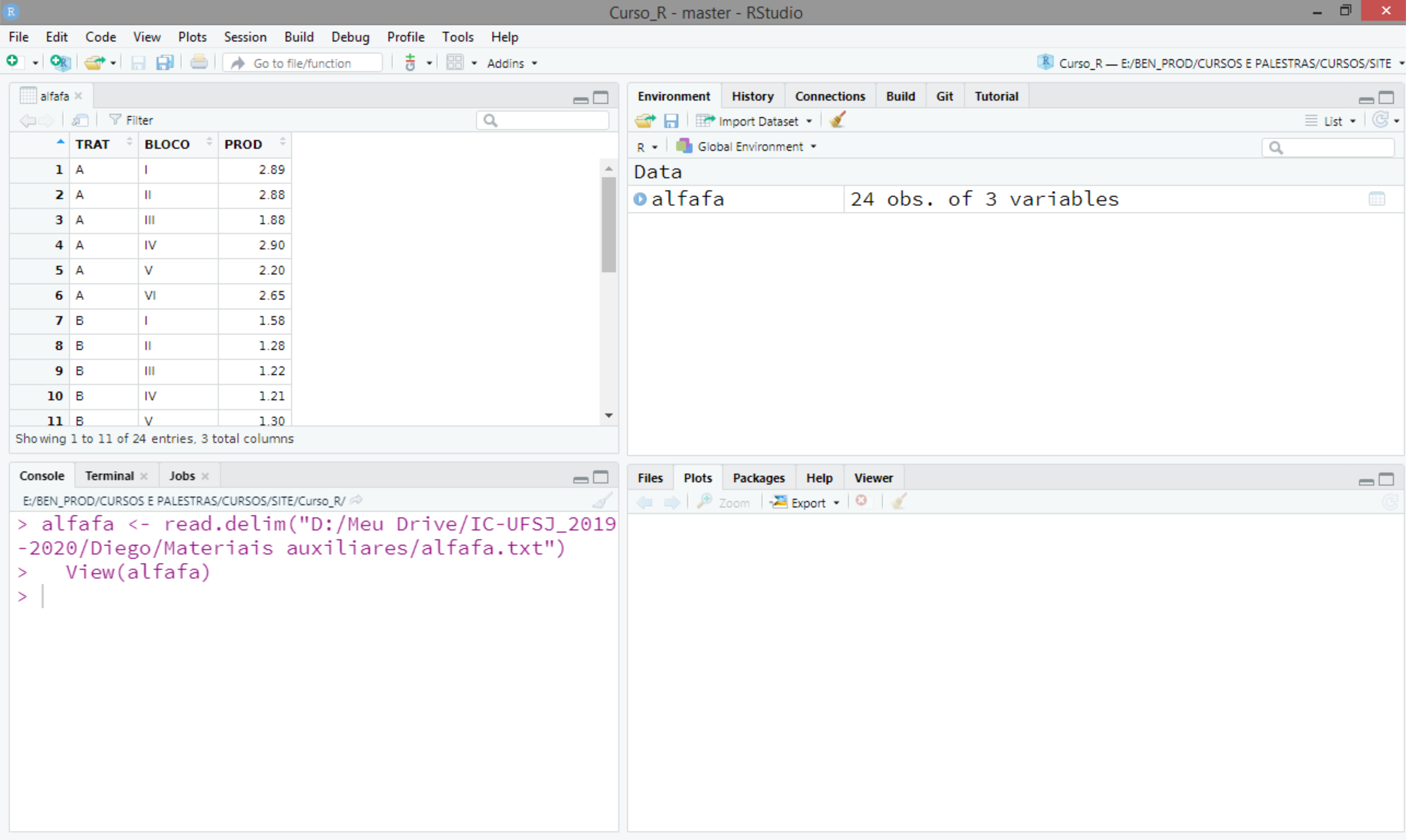
Figure 16: Usando o RStudio para importar dados.
A outra forma é utilizar linhas de comando. Para isso utilizaremos a função read.table(). Antes de importarmos o banco de dados, algo interessante é inserir o arquivo de dados no diretório de trabalho no ambiente R. Para verificar o ambiente de trabalho use a função getwd(). Para alterar o local do ambiente de trabalho use setwd(). Se esse procedimento não for realizado, o usuário deve informar na função read.table(), o local exato do arquivo de texto.
Vamos usar como diretório o local C:\cursor. Lembre-se que no R, a barra deve ser invertida. Vamos inserir nesse diretório três arquivos alfafa.txt, datast1980.txt e producao.csv.
Os três conjuntos de dados são:
- alfafa.txt
| TRAT | BLOCO | PROD |
|---|---|---|
| A | I | 2.89 |
| A | II | 2.88 |
| A | III | 1.88 |
| A | IV | 2.90 |
| A | V | 2.20 |
| A | VI | 2.65 |
| B | I | 1.58 |
| B | II | 1.28 |
| B | III | 1.22 |
| B | IV | 1.21 |
| B | V | 1.30 |
| B | VI | 1.66 |
| C | I | 2.29 |
| C | II | 2.98 |
| C | III | 1.55 |
| C | IV | 1.95 |
| C | V | 1.15 |
| C | VI | 1.12 |
| D | I | 2.56 |
| D | II | 2.00 |
| D | III | 1.82 |
| D | IV | 2.20 |
| D | V | 1.33 |
| D | VI | 1.00 |
- datast1980.txt
| trt | y |
|---|---|
| 1 | 19.4 |
| 1 | 32.6 |
| 1 | 27.0 |
| 1 | 32.1 |
| 1 | 33.0 |
| 2 | 17.7 |
| 2 | 24.8 |
| 2 | 27.9 |
| 2 | 25.2 |
| 2 | 24.3 |
| 3 | 17.0 |
| 3 | 19.4 |
| 3 | 9.1 |
| 3 | 11.9 |
| 3 | 15.8 |
| 4 | 20.7 |
| 4 | 21.0 |
| 4 | 20.5 |
| 4 | 18.8 |
| 4 | 18.6 |
| 5 | 14.3 |
| 5 | 14.4 |
| 5 | 11.8 |
| 5 | 11.6 |
| 5 | 14.2 |
| 6 | 17.3 |
| 6 | 19.4 |
| 6 | 19.1 |
| 6 | 16.9 |
| 6 | 20.8 |
- producao.csv
| x | y |
|---|---|
| 1 | 6.7 |
| 2 | 7.9 |
| 3 | 9.1 |
| 4 | 6.6 |
| 5 | 7.5 |
| 6 | 8.8 |
| 7 | 7.7 |
| 8 | 7.6 |
| 9 | 6.5 |
| 10 | 7.9 |
| 11 | 8.7 |
| 12 | 6.2 |
| 13 | 7.9 |
| 14 | 7.4 |
| 15 | 9.7 |
| 16 | 6.2 |
| 17 | 4.9 |
| 18 | 5.6 |
| 19 | 7.0 |
| 20 | 6.0 |
Vejamos as linhas de comando para importar os dados.
# Diretorio
getwd()
# Mudadando para o diretorio de interesse
setwd("C:/cursor")
# Verificando os arquivos no diretorio de trabalho
list.files()
# Importando os dados apontando para o diretorio do arquivo
dados1 <- read.table(file = "C:/cursor/alfafa.txt", header = TRUE)
# Considerando que o arquivo esta no diretorio de
# trabalho, isto eh, getwd()
dados2 <- read.table("alfafa.txt", header = TRUE)
# Importando os dados com decimais com ',' apontando para o diretorio do arquivo
dados3 <- read.table(file = "C:/cursor/dadost1980.txt", header = TRUE, dec = ",")
# Considerando que o arquivo esta no diretorio de
# trabalho, isto eh, getwd()
dados4 <- read.table(file = "dadost1980.txt", header = TRUE, dec = ",")
# Importando os dados com decimais ',', e separados por ';' apontando para o diretorio do arquivo
dados5 <- read.table(file = "C:/cursor/producao.csv", header = TRUE, dec = ",", sep = ";")
# Considerando que o arquivo esta no diretorio de
# trabalho, isto eh, getwd()
dados6 <- read.table(file = "producao.csv", header = TRUE, dec = ",", sep = ";")
# Importando da internet
dados7 <- read.table(file = "https://bendeivide.github.io/cursor/dados/alfafa.txt", header = TRUE)
Na última linha de comando, mostramos que também é possível importar dados de arquivos de texto da internet, e claro considerando que o usuário está com acesso a internet no momento da importação. E um recurso interessante que pode ser feito, principalmente para este caso, é salvar o banco de dados em um arquivo de dados no .RData. Dessa forma, todos os dados, inclusive os importados da internet serão agora armazenados nesse tipo de arquivo, e não precisaremos, nesse caso, de acesso a internet. Para salvar, usamos a função save(). Para carregar os dados e armazená-lo no ambiente global, usamos a função load(). Vejamos,
# Diretorio
getwd()
# Verificando os arquivos do diretorio de trabalho
list.files()
# Importando os dados da internet
dados7 <- read.table(file = "https://bendeivide.github.io/cursor/dados/alfafa.txt", header = TRUE)
# Salvando em '.RData'
save(dados7, file = "alfafa.RData")
# Carregando '.RData' para o ambiente global
load("alfafa.RData")
Percebemos que as extensões .txt e .csv são idênticos, exceto pela estrutura de como os dados estão dispostos. Para comprovar isso, o usuário manualmente poderá mudar a entensão de um arquivo do tipo .csv para um arquivo .txt e observar em um bloco de notas.
Até esse momento do curso, usamos as funções no R sem apresentar os argumentos dessas funções dentro dos parênteses. Isso porque quando inserimos os valores dos argumentos na posição correta destes, não precisaremos inserir o nome dos argumentos. Por exemplo, já usamos anteriormente a função mean() que calcula a média de um conjunto de valores, por exemplo, valores <- 1:10. Temos como primeiro argumento para essa função o x que representa um objeto R que recebe os valores para o cálculo. Assim, como sabemos que x é o primeiro argumento dessa função, podemos omitir o seu nome e calcular a média por mean(valores), que é o mesmo que mean(x = valores). Para mais detalhes, ?mean(). Para mais detalhes sobre a função read.table(), use ?read.table().
Funções
Mais uma vez, nos reportamos aos princípios do R (Chambers (2016)), mais especificamente ao segundo princípio,
- Princípio da função: Tudo que acontece no R é uma chamada de função.
Para que possamos compreender esse princípio, precisamos entender que uma função é um objeto, isto é, modo function18, assim como os vetores são, seguindo as mesmas ideias comentadas na seção anterior, e que as funções podem ser dividas em três componentes:
- Argumentos, função
formals(), - Corpo, função
body()e - Ambiente, função
environment().
Para o caso das funções primitivas, escritas na linguagem C, essa regra foge a excessão, e será comentado nos módulos seguintes. Dizemos que funções são primitivas de modo builtin ou special.
A ideia de função aqui não pensando como uma relação matemática, mas como um sistema que tem uma entrada e saída. Podemos ter funções no ambiente R que organiza dados, e não operações matemáticas por exemplo. Vejamos a função sort() do pacote base que ordena de forma crescente ou descrescente um conjunto de valores,
# Vetor
y <- c(5, 3, 4); y
[1] 5 3 4# Funcao
sort(x = y)
[1] 3 4 5# Argumentos da funcao sort
formals(sort)
$x
$decreasing
[1] FALSE
$...# Corpo da funcao
body(sort)
{
if (!is.logical(decreasing) || length(decreasing) != 1L)
stop("'decreasing' must be a length-1 logical vector.\nDid you intend to set 'partial'?")
UseMethod("sort")
}# Ambiente
environment(sort)
<environment: namespace:base>Nesse caso, os argumentos x, decreascing e ..., são nomes que aguardam receber objetos para a execução da função sort(). Nem todos os argumentos necessitam receber objetos, a estes chamamos de argumentos padrão, como o caso do argumento decreascing com padrão igual a FALSE, que significa que o ordenamento dos dados será de forma não-decrescente. Observe que na função sort() entramos apenas com o argumento x = y, não precisando inserir decreascing = FALSE. Agora, para modificar o argumento padrão, basta acrescentar a alteração na função, isto é,
# Funcao
sort(x = y, decreasing = TRUE)
[1] 5 4 3O '...' é um argumento especial e significa que pode conter qualquer número de argumentos. Geralmente é utilizado em uma função quando não se sabe o número exato de argumentos. Na subseção Como criar funções, entenderemos melhor.
O próximo item é o corpo da função. É nele que inserirmos as instruções, isto é, as linhas de comandos necessárias a que destina a sua criação. Uma outra forma de acessarmos o corpo das funções é digitar no console apenas o seu nome sem o parêntese, isto é, sort.
Por fim, o ambiente que no caso da função sort() representa o ambiente do pacote base, isto é, o namespace do pacote.
Podemos observar que essas funções utilizadas até agora, não foram criadas pelo usuário. Estas funções vieram do que chamamos de pacotes. Alguns pacotes estão disponíveis quando instalamos o R, dizemos que estes são os pacotes da Base do R, para a linguagem. O principal pacote deles é o pacote base. Os demais pacotes desenvolvidos podem ser obtidos via CRAN, e falaremos mais adiante.
O ambiente R apresenta uma versatilidade de manuais para a linguagem. Por exemplo, para verificar informações sobre um determinado pacote como o base, use help(package = "base"). A função help pode ser utilizada para funções de pacotes anexados. Por exemplo, help("sort"). Uma outra função que pode ser usada para procurar por funções com determinado parte de nome é apropos(), isto é, para o exemplo anterior, temos apropost("sort"). O pacote base sempre estará anexado, isto é, disponível no caminho de busca para a utilização. Para os que não estão anexados, a função help deve informar o nome da função que necessita de ajuda, bem como o seu pacote. Por exemplo, temos uma função read.dbf() do pacote foreign, Base do R, porém, ele não está anexado19 ao inicializar o R. Assim, caso não anexe o pacote, usando library("foreign"), a ajuda sobre a função pode ser realizada com help("read.dbf", package = "foreign"). Outras sintaxes para a função help() é usar ? antes do nome de uma função de um pacote anexado, isto é, ?sort().
Para os manuais de ajuda na internet, use ?? antes do nome da função, por exemplo ??sort(), read.dbf(), etc. Essa sintaxe não precisa dos pacotes estarem anexados para ajuda de determinada função, porém o pacote necessita estar instalado. Para mais detalhes sobre pacotes, teremos uma seção sobre Pacotes.
As chamadas de funções podem ocorrer de três formas: aninhada, intermediária ou de forma pipe. As duas primeiras formas da Base do R, e o segundo foi uma sintaxe utilizada pelo pacote magrittr, e na versão do R 4.1, já existe o operador pipe nativo (|>), sendo comentado no módulo Programação em R (Nível Intermediário).
Suponha que desejamos calcular o desvio padrão de um conjunto de valores. Vamos utilizar as três formas:
- Aninhado:
# Gerando 100 numeros aleatorios de uma distribuicao normal
set.seed(10) # Semente
x <- rnorm(100)
# Calculando o desvio padrao
sqrt(var(x))
[1] 0.9412359- intermediária:
- Pipe:
[1] 0.9412359A ideia da chamada de função aninhada é inserir função como argumento de funções sem necessidade de associar nomes aos objetos. A ordem de execução começa sempre da direita para a esquerda. No caso da chamada de função intermediária, associamos nomes a cada função, e os passos seguem. Por fim, o operador especial pipe (%>%) tem como primeiro operando o primeiro argumento da função no segundo operando.
Quando desenvolvemos pacotes, preferimos os dois primeiros, pois é a forma tradicional de chamadas de função no R. A chamada de função pipe é muito utilizada para ciência dos dados.
Estruturas de controle
As funções que utilizaremos, a seguir, são utilizadas quando desejamos realizar processos repetitivos para um determinado fim ou condicionado, as famosas estruturas de controle. Assim, como em outras linguagens, as funções utilizadas são: if(), switch(), ifelse(), while(), repeat, for(). Esses objetos tem modo special20, porque as expressão não são necessariamente avaliadas. Já a função ifelse() é de modo closure21. Esse é um tipo de função implementado por function().
A ideias das estruturas de controle de modo special é:
função (condição) {
expressão
}A sintaxe das funções repeat e swicth() fogem um pouco desse padrão, e explicaremos em sua aplicação.
Apesar da linguagem R ser interpretada, acaba ganhando a fama de que as funções loops são mais lentas. Em alguns casos, a construção do algoritmo proporciona isso e não a implementação dessas funções em si. Um exemplo que podemos citar, são as cópias de objetos podem ter um gasto de memória ativa imenso no processo e proporcionar um gasto computacional. E isso foi devido a forma de como o algoritmo copiou os objetos, e não as funções lopps. Veremos isso no módulo Programação em R (Nível Intermediário).
A primeira estrutura é o if(), com sintaxe:
if (condição) {
expressão sob condição = TRUE
} else {
expressão sob condição = FALSE
}ou de forma alternativa:
if (condição) {
expressão sob condição = TRUE
}Observe o primeiro exemplo,
# Objeto
i <- 5
# Estrutura if()
if (i > 3) {
print("Maior que 3!")
}
[1] "Maior que 3!"Vejamos mais um exemplo,
# Objeto numerico
x <- 10
# Estrutura 'if'
if (is.numeric(x)) {
print("Isso é um número")
} else {
print("Isso não é um número")
}
[1] "Isso é um número"# eh o mesmo que
if (is.numeric(x) == TRUE) {
print("Isso é um número")
} else {
print("Isso não é um número")
}
[1] "Isso é um número"observe na primeira forma que a condição na função nós não precisamos perguntar se is.numeric(x) == TRUE, porque isso já é implícito na função, e acaba levando a primeira expressão print("Isso é um número"). No R, essa estrutura de controle não é vetorizado, isto é, se a condição houver um vetor lógico maior que 1, apenas os primeiros itens serão usados. Vejamos,
# Objetos
x <- 5
w <- 3:8
# Primeira sintaxe (Preferivel)
if (x < w) {
x
} else {
w
}
[1] 3 4 5 6 7 8# Segunda forma
# if (x < w) x else w
Como forma alternativa, pode ser usado a função ifelse, cuja sintaxe é:
ifelse(condição, expressão sob TRUE, expressão sob FALSE)Assim, temos:
# Objetos
x <- 5
w <- 3:8
# Primeira sintaxe (Preferivel)
ifelse(x < w, x, w)
[1] 3 4 5 5 5 5Podemos estar interessados em resultados específicos para determinadas condições. Por exemplo,
x <- 2 # numero ou palavra
# Estrutura 'if'
if (is.character(x)) {
"palavra"
} else {
if ((x %% 2) == 0) {
"numero_par"
} else {
if ((x %% 2) == 1) {
"numero_impar"
}
}
}
[1] "numero_par"Outro exemplo seria,
# Objeto
set.seed(15) # Fixando a semente
x <- rnorm(1000) # Gerando 1000 numeros aleatorios
# medida descritiva
opcao <- "media" # opcoes: "media", "mediana", "medapar" (media aparada)
if (opcao == "media") {
cat("A média aritmética é:", round(mean(x), 4))
} else {
if (opcao == "mediana") {
cat("A mediana é:", round(mean(x), 4))
} else {
if (opcao == "medapar") {
cat("A média aparada é:", round(mean(x, trim = 0.1), 4))
}
}
}
A média aritmética é: 0.037Poderíamos utilizar a função switch(), para esse mesmo código, isto é,
switch(opcao,
media = cat("A média aritmética é:", round(mean(x), 4)),
mediana = cat("A mediana é:", round(mean(x), 4)),
medapar = cat("A média aparada é:", round(mean(x, trim = 0.1), 4))
)
A média aritmética é: 0.037As três estruturas de loop no R são: repeat, while() e for(). A primeira delas é repeat, apresentamos sua sintaxe:
repeat {
expressão ...
}Juntamente com o repeat, usamos as funções break e next, pois a função repeat não tem uma condição explícita. Vejamos, alguns exemplos.
# Contador
i <- 1
# Loop repeat
repeat {
if (i > 5) {
break
} else {
print(i)
i <- i + 1
}
}
[1] 1
[1] 2
[1] 3
[1] 4
[1] 5Observe que quando i <- 6, o ciclo entrou na condição if(i > 5) e avaliou a função break, que resultou na quebra do ciclo. Assim, a função print(i) não foi mais acionada, e os resultados foram impressos até o número 5. No próximo exemplo, acrescentamos a função next.
# Contador
i <- 1
# Loop repeat
repeat {
if (i > 5) {
break
}
else {
if (i == 3) {
i <- i + 1
next
}
print(i)
i <- i + 1
}
}
[1] 1
[1] 2
[1] 4
[1] 5Similar a essa função, temos while(), que agora, temos uma condição de parada, cuja sintaxe é:
while (condição) {
expressão ...
}Nessa função, também podemos usar as funções break e next, como usados na função repeat. Vejamos um exemplo,
# Contador
i <- 1
# Loop while
while (i <= 5) {
print(i)
i <- i + 1
}
[1] 1
[1] 2
[1] 3
[1] 4
[1] 5Nesse caso, não foi necessário utilizar a função break, devido a condição que ela permite impor ao ciclo. Vejamos um outro exemplo,
# Contador
i <- 1
# Loop while
while (i <= 5) {
if (i == 3) {
i <- i + 1
next
}
print(i)
i <- i + 1
}
[1] 1
[1] 2
[1] 4
[1] 5Observamos a utilização da função next, similar ao que foi realizado com a função repeat. Por fim, a função for(), cuja sintaxe é dada por:
for (contador in lista) {
expressão ...
}Com essa função, também podemos utilizar break e next. Porém, o controle do ciclo nessa sitação é maior, e menos necessário essas funções. Vejamos um primeiro exemplo,
# Loop for
for (i in 1:5) {
print(i)
}
[1] 1
[1] 2
[1] 3
[1] 4
[1] 5Observe que a implementação, dos algoritmos anteriores, é bem mais simples usando a função for(). Vejamos um próximo exemplo,
# Loop for
for (i in 1:5) {
if (i == 3) {
next
}
print(i)
}
[1] 1
[1] 2
[1] 4
[1] 5Mais uma vez, foi utilizado a função next, sem necessidade agora, de atualizar o contador i, uma vez que já está definido na função for().
Veremos na próxima subseção, que temos funções de alto nível que podem substituir as funções loops. São as chamadas funções da família apply.
Como criar funções
Até esse momento, usamos funções já desenvolvidas no R, seja dos pacotes da Base, seja via instalação dos pacotes via CRAN. Agora, iremos desenvolver as nossas próprias funções.
Como falado anteriormente, no início dessa seção, a estrutura da função criada se mantém, argumento, corpo e ambiente. Para isso, usaremos a função function. O modo desse objeto é closure22. Vejamos a sua sintaxe,
# Forma usual
nome_funcao <- function(arg1, arg2, ...) {
corpo: comandos..
}
# Forma simplificada
nome_funcao <- function(arg1, arg2, ...) corpoVejamos um primeiro exemplo,
fun1 <- function(x) {
res <- x + 1
return(res)
}
Nesse caso, temos uma função chamada fun1, cujo argumento de entrada é x. Observe que uma função é como um objeto do tipo vetor, associamos um nome ao objeto da mesma forma. O corpo apresenta uma delimitação por chaves {...}, em que apresenta um comando de atribuição res que se associa a soma x + 1, e por fim, o resultado dessa função, imprime res, por meio da função return(). Para utilizar essa função, fazemos
fun1(x = 5)
[1] 6O que aconteceu foi que ao assumir x = 5, essa informação foi repassada para o corpo da função, aonde existia x, foi substituído por 5, para esse caso. A função + é chamada, a expressão x + 1 avaliada, e nome res se associa ao resultado da expressão. Por fim, quando a função fun1 é chamada, o resultado de res é impresso no console, por meio de return(res). Vamos verificar os três componentes da função fun1,
# Argumentos
formals(fun1)
$x# Corpo
body(fun1)
{
res <- x + 1
return(res)
}# Ambiente
environment(fun1)
<environment: R_GlobalEnv>O corpo da função é executado de forma sequencial, a partir da primeira linha até a última. Apesar de recomendado, também não é obrigatório o uso da função return(), veja
# Funcao
fun2 <- function(x) x + 1
# Executando
fun2(5)
[1] 6Função anônima
Podemos também ter o que chamamos de função anônima, da qual não associamos nome as funções. Contudo, sendo criada esta não pode ser recuperada como qualquer outro objeto. Essa forma é interessante quando não precisamos dela após o seu uso. Por exemplo, queremos calcular a integral, \[ \int^{1}_{0}x^2dx = \frac{1}{3}, \]
e criamos uma função \(x^2\). Então,
integrate(f = function(x) x^2,
lower = 0,
upper = 1)
0.3333333 with absolute error < 3.7e-15A função integrate é utilizada para o cálculo de integral, do qual, passamos os argumentos, função (f), limite inferior (lower) e limite superior (upper) da integração, respectivamente. Observe que não houve necessidade de nomear a função no argumento, pois não há objetivo de reutilizado.
Chamadas de função
As chamadas para essas funções ocorrem da mesma forma que as funções apresentadas da Base do R. Vejamos o cálculo do desvio padrão novamente,
# Funcao auxiliar 1
aux1 <- function(x) x - mean(x)
# Funcao auxiliar 2
aux2 <- function(x) x^2
# Funcao auxiliar 3
aux3 <- function(x) {
sum(x) / (length(x) - 1)
}
# Gerando 100 numeros aleatorios de uma distribuicao normal
set.seed(10)
x <- rnorm(100)
# Calculo do desvio padrao (aninhado)
sqrt(aux3(aux2(aux1(x))))
[1] 0.9412359# Calculo do desvio padrao (intemediario)
dp <- aux1(x)
dp <- aux2(dp)
dp <- aux3(dp)
dp <- sqrt(dp)
dp
[1] 0.9412359# Calculo do desvio padrao (pipe)
x %>%
aux1() %>%
aux2() %>%
aux3() %>%
sqrt()
[1] 0.9412359Ordenação de argumentos
Os argumentos nas funções podem ser nomeados ou não. Quando nomeados, a ordem como são inseridos na função não importa. Já os argumentos não nomeados, seus valores precisam estar na ordem como a função foi desenvolvida. Vejamos,
estdesc <- function(x, opcao) {
res <- switch(opcao,
media = round(mean(x), 4),
mediana = round(mean(x), 4),
medapar = round(mean(x, trim = 0.1), 4))
return(res)
}
# Objeto
set.seed(15)
x <- rnorm(1000)
# Argumentos nomeados na funcao
estdesc(x = x, opcao = "media")
[1] 0.037estdesc(opcao = "media", x = x)
[1] 0.037# Argumentos não nomeados ordenados
estdesc(x, "media")
[1] 0.037# Argumentos não ordenados (Gera erro)
estdesc("media", x)
Error in switch(opcao, media = round(mean(x), 4), mediana = round(mean(x), : EXPR deve ser um vetor de comprimento 1Objeto reticências (...)
O objeto reticências é do tipo pairlist, um tipo de objeto usado bastante internamente no R, e dificilmente utilizado no código interpretado. De modo que não temos acesso direto ao objeto .... Contudo, esse objeto tem um papel fundamental nas funções, quando damos a liberdade de inserir mais argumentos além dos definidos na função. Vejamos,
# Funcao que plota um grafico
grafico <- function(x, y, ...) {
plot(x = x, y = y, ...)
}
# Vetores
x <- 1:10; y <- rnorm(10)
# Chamada 1, com os argumentos definidos
grafico(x = x, y = y)
# Chamada 2, inserindo argumentos nao definidos
grafico(x = x, y = y, main = "Título")
Isso ocorre, porque temos a função plot() no corpo da função grafico() que apresenta muitos argumentos. Dessa forma, criamos a função grafico(), com os argumentos que representam as coordenadas, mas a reticências (...) garantem que os demais argumentos de plots, omitidos, possam ser utilizados.
Escopo léxico
Vamos retornar aos componentes da função fun1,
# Argumentos
formals(fun1)
$x# Corpo
body(fun1)
{
res <- x + 1
return(res)
}# Ambiente
environment(fun1)
<environment: R_GlobalEnv>Observe que o último componente é o ambiente onde o nome fun1 foi associado a função. Este ambiente é chamado de ambiente envolvente. Nesse caso é o ambiente global. Contudo, quando a função é executada, momentaneamente é criado o ambiente de execução. É nesse ambiente que os nomes que estão no corpo da função são associados aos objetos. Vejamos um primeiro exemplo,
x <- 10
fun <- function() {
x <- 2
x
}
# Chamando a funcao fun
fun()
[1] 2Por causa do ambiente de execução que o objeto x dentro da função é retornado, ao invés do que foi definido fora da função. Isso porque o ambiente de execução mascara os nomes definidos dentro da função dos nomes definidos fora da função. Esse é uma primeira característica do escopo léxico nas funções em R.
Anteriormente, falamos sobre a atribuição, que representa a forma como os nomes se associam aos objetos. Agora, o escopo vem a ser a forma como os nomes encontram seus valores associados. O termo léxico significa que as funções podem encontrar nomes e seus respectivos valores associados, definidos no ambiente onde a função foi definida, isto é, no ambiente de função. Claro que isso segue regras, e a primeira foi a máscara de nome falada anteriormente.
Porém quando não existe um nome vinculado a um objeto, e este foi definido no ambiente de função, o valor é repassado para o corpo da função. Observe,
x <- 10
fun <- function() {
x
}
# Chamando a funcao fun
fun()
[1] 10O resultado de fun() foi 10, porque como a função procurou no ambiente de execuções e não encontrou esse nome, a função foi até o ambiente superior, no caso, o ambiente de execuções. Como falado anteriormente, todo ambiente tem um pai (ou ambiente superior). Essa hierarquização é observada no caminho de busca, que pode ser acessado por search(), ou seja,
search()
[1] ".GlobalEnv" "package:magrittr" "package:leaflet"
[4] "package:stats" "package:graphics" "package:grDevices"
[7] "package:utils" "package:datasets" "package:methods"
[10] "Autoloads" "package:base" O ambiente corrente do R sempre será o ambiente ambiente global (.GlobalEnv). O ambiente de execução não aparece, porque ele é momentâneo. Então após buscar no ambiente de execução e não encontrar, é pelo caminho de busca que a função irá procurar pelos objetos inseridos no corpo da função. Ele só existe quando a função é chamada e não quando ela é definida, isto é,
# Funcao
fun <- function() x + 10
# Objeto 1
x <- 10
# Chamada1
fun()
[1] 20# Objeto 2
x <- 20
# Chamada 2
fun()
[1] 30Observe que após ser criada a função fun(), o nome x se associou ao objeto 10. Posteriormente, fun() foi chamada e o resultado foi 20. Contudo, o nome x se associou a outro objeto 20, e após a segunda chamada da função fun(), o resultado foi 30, porque a função procura os valores quando ela é executada, e não quando é criada, é a característica de pesquisa dinâmica do escopo léxico.
Uma outra situação é a seguinte,
# Objeto
n <- 1
# Funcao
fun <- function() {
n <- n + 1
n
}
# Chamada 1
fun()
[1] 2# Chamada 2
fun()
[1] 2Nessas linhas de comando, poderíamos pensar que após ter executado a primeira chamada, que retornou valor 2, a segunda chamada retornaria o valor 3, como ocorre com as variáveis estáticas na linguagem C. Aqui nesse caso, resultado independente do número de chamadas será sempre o mesmo, porque uma outra característica do escopo léxico no R é o novo começo, porque a cada vez que a função é executada um novo ambiente de execução é criado, e portanto, cada execução dos comandos de atribuição e expressão são executados de forma independentes nas chamadas de funções.
Algo que não havíamos falado anteriormente, é que a função function() permite que não seja definido argumentos, devido a flexibilidade do escopo léxico das funções em R. É essa característica que faz com que os comandos no corpo das funções encontrem os objetos que não estão definidos na própria função. Mais detalhes, será visto no módulo Programação em R (Nível Intermediário).
Boas práticas de como escrever um código
Nesse momento, entendemos os principais objetos para escrevermos os nossos scripts. Quando escrevemos um código, duas consequências ocorrem: - guardá-lo para futuras consultas, ou - compartilhamento.
Nesses dois casos, percebemos que alguém irá ler esse código, ou até mesmo o próprio usuário, irá retornar àquelas linhas de código e tentar raciocinar quais as ideias por trás disso tudo. Para um melhor entendimento de seu script, nada mais importante do que uma boa escrita, separação das estruturas por hierarquização, comentários, etc.
Uma primeira ferramenta que pode ser configurada para quem usa o rstudio é acionar todas as opções de diagnóstico do seu código. Para isso no menu:
Tools > Global options > Code > Editing. Marque todas as opções emGeneral;Tools > Global options > Code > Display, Marque todas as opções;Tools > Global options > Code > Diagnostics. Marque todas as opções emR Diagnostics.
Com isso, colorações nas linhas de comando ocorrerão, distinguindo diversas estruturas, como linhas de comentário, funções, espaçamentos, dentre outras coisas.
Uma vez feito isso, vamos para o passo seguinte que são as boas práticas de como se escrever um script. Temos algumas ferramentas prontas, como o pacote styler e como alternativa o formatR, que automatiza todo o nosso código seja em script, contido em um pacote, ou diretório. Acesse https://yihui.org/formatr, para mais detalhes. Para instalar e anexar o pacote styler, use as linhas de comando:
# Instalando pacote
install.packages(styler)
# Carregando e anexando
library(styler)
Vejamos a seguinte ilustração, para entendermos a funcionabilidade desse pacote .
.](images/stylecode.gif)
Figure 17: Estilo de código com o pacote styler.
Pela Figura 17, percebemos que inicialmente, a leitura do código sem nenhum padrão de hierarquização, de funções dentro de funções, torna mais difícil a leitura quando não seguimos algumas regras básicas de espaçamentos iniciais nas linhas de comando. Ao passo que, após aplicar a função styler:::style_active_file(), conseguimos distinguir quais as funções que estão dentro de outras, bem como as suas expressões.
Nomes de arquivos
Apesar de toda essa facilidade, vamos apresentar algumas ideias. A primeira noção é o nome do arquivo. Padronizem a extensão .R e evitem espaços em nomes compostos,
Boa escolha:
-----------
script.R
nome_composto.R
nome-composto.R
Má escolha:
-----------
script.r
nome composto.rComentar as linhas de comando
Sempre que escrever uma linha de comando ou uma sequência de linhas de comando para um mesmo fim, comente sobre a ideia que esse fragmento de código quer repassar, usando #. Evitem acentos e símbolos especiais, porque os novos acessos ao script, dependendo da codificação que o RStudio esteja utilizando ou o seu sistema operacional, pode desconfigurar todo o arquivo.
Boa escolha:
-----------
# Objeto x
x <- 1:10
# Calculo da media
mean(x)
Má escolha:
-----------
x <- 1:10
mean(x)Nomes de objetos
Levem em consideração que nomes de objetos é uma forma de acessarmos os objetos no R, e não os nomes das variáveis que serão utilizados em nossos relatórios para escrever sobre um problema real. Portanto, a escolha desses nomes devem ser mais simples, curtos, sem acentos ou símbolos não ASCII, e de preferência apenas letras minúsculas.
Boa escolha:
-----------
nome_curto
aux1
Má escolha:
-----------
nome_muito_grande
Nome_Grande
Aux1Sempre que possível evite nomes já utilizados no R, para outros fins,
Má escolha:
-----------
T <- "Nada"
c <- 5
sd <- 5 + 1
mean <- 3 * 4Para verificar se determinado nome já existe, use a função exists(), isto é, exists("mean"). Se a função retornar TRUE, significa que já existe esse nome, caso contrário, FALSE.
Sintaxe
Um dos erros mais comuns na sintaxe de um código é o espaçamento entre os operadores básicos na linguagem R, exceto para os operadores :, :: e :::. Use sempre um espaço após a vírgula.
Boa escolha:
-----------
x <- 1:10
media <- mean(x + 1 / length(x), na.rm = TRUE)
base::mean(x)
Má escolha:
-----------
x <- 1 : 10
media<-mean(x+1/length(x),na.rm=TRUE)
base :: mean(x)Para facilitar pode ser utilizado a função styler::style_text(), isto é,
comando <- "media<-mean(x+1/length(x),na.rm=TRUE)"
styler::style_text(comando)
media <- mean(x + 1 / length(x), na.rm = TRUE)Se necessário use mais de um espaço em uma linha para o alinhamento para <- ou =, bem como um espaço antes do parêntese, a menos que seja uma função,
Boa escolha:
-----------
data.frame(a = 1,
b = "Ben")
function(){
x <- 10
vari <- x + 1
return(vari)
}
for (i in 1:10) i + 1
Má escolha:
-----------
function () 1
for(i in 1:10) i + 1Não insira espaços na condição entre parêntese ou nos itens de indexação, a menos que este último contenha uma vírgula,
Boa escolha:
-----------
if (verbose)
x11 <- mat[1, 1]
x1 <- mat[1, ]
Má escolha:
-----------
if ( verbose )
x11 <- mat[1,1]
x1 <- mat[1,]Quando usamos chaves em um comando, devemos evitar abri-lo e fechá-lo na mesma linha. E ainda, quando é função, as linhas de comando inseridas dentro das chaves, inserimos um recuo de dois espaços para entendermos a hierarquização das funções, isto é, função dentro de função. Veja,
Boa escolha:
-----------
fx <- function(x) {
if (x > 2) {
print("Maior que 2!")
} else {
print("Menor que 2!")
}
}
for (i in 1:10) {
x <- i + 1
}
Má escolha:
-----------
fx <- function(x) {
if (x > 2) {
print("Maior que 2!")
} else {
print("Menor que 2!")
}
}
for (i in 1:10) {x <- i + 1}Observe também que sempre quando iniciamos a abertura de uma chave, damos um espaço para separar do fechamento do parêntese, sempre quando usamos em uma função. Por fim, sempre use a atribuição com <-, e = para definir os argumentos de uma função,
Boa escolha:
-----------
fx <- function(x = NULL) 10
a <- "Nome"
Má escolha:
-----------
fx = function(x = NULL) 10
a = "Nome"Para códigos mais complexos, use o pacote styler e como alternativa o formatR, pois muito mais recursos podem ser utilizados. Para consultas, acesse também Wickham (2015), mais específico, Estilo de código, e ainda Advanced R, 1ed, Style guide, The tidyverse style guide e Google’s R Style Guide.
Pacotes
O pacote em R é um diretório de arquivos necessários para carregar um código de funções, dados, documentações de ajuda, testes, etc. O próprio R em sua instalação, contém 30 pacotes, que dizemos que são as funções mínimas para a utilização da linguagem. Nesse diretório não há apenas códigos em R, mas um pacote fonte (do inglês, source package), contendo os arquivos mencionados acima, ou um arquivo compactado, de extensão .tar.gz do pacote fonte, ou um pacote instalado, resultado da função R CMD INSTALL, que será visto no módulo Programação em R (Nível Avançado). Isso acontece no SO Linux. Para as plataformas Windows e Macintosh, existem também os pacotes binários ou compactados com a extensão .zip ou .tar.gz.
Um pacote, portanto é a unidade básica para o compartilhamento de um código. Atualmente, até 11/03/2021, o número de pacotes disponíveis é 17.295, e isso no CRAN, isto é, na Comprehenive R Archive Network, o repositório oficial de pacotes no R. Qualquer usuário pode publicar um pacote e disponibilizá-lo sob o CRAN. Para isso, uma série de testes iniciais são realizados para verificar se não há problemas. Significa também se um pacote está disponível no CRAN ele funcionará nas três plataformas mais usadas em sistema operacional, SO Linux ou sitemas Unix, SO Windows e SO Macintosh. Há outros repositórios que podem ser disponibilizados os pacotes, como por exemplo, no GitHub. Porém, não há a garantia de que possa estar funcionando corretamento em seu sistema operacional, como os pacotes disponíveis no CRAN.
Para uma instalação mais rápida no CRAN, há espelhos disponíveis no Brasil, do qual o primeiro foi desenvolvido e está funcionando até hoje na UFPR.
Contudo, uma coisa deve ficar claro, que erroneamente, alguns usuários chamam pacote de biblioteca. Nas documentações do R, biblioteca é o diretório onde os pacotes são instalados, também chamados de diretório de biblioteca ou diretório de arvores. O outro sentido de biblioteca é o de biblioteca compartilhada (dinâmica ou estática), que armazenam código compilado que se vinculam aos pacotes, por exemplo, no Windows são as DLLs.
A estrutura básica de um pacote é apresentada na Figura 18.
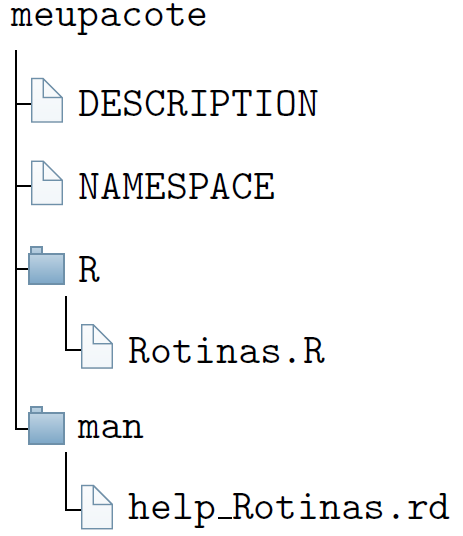
Figure 18: Esqueleto básico de um pacote.
Vejamos as ideias básicas desses subdiretórios e arquivos,
- DESCRIPTION: Esse é um arquivo de texto, contendo informações básicas como o título do pacote, versão, licença, descrição, nome dos autores, e o mantenedor do pacote, isto é, para quando um pacote estiver com problema ou o CRAN entre em contato, será para este último. Esses as informações obrigatórias que devem ter nesse arquivo.
- NAMESPACE: Esse arquivo embora tenha tenha muita semelhança com a linguagem R, o seu conteúdo, se destina a importação e exportação de funções no pacote. Será nesse arquivo, que diremos quais os pacotes que ele depende, isto é, as funções, e quais as funções exportadas, visíveis, que devem ser apresentadas aos usuários.
- R/: Esse subdiretório apresenta os scripts com as funções em R. é o cérebro do pacote.
- man/: Esse subdiretório apresenta os arquivos de ajuda, com extensão .Rd. Isso significa, que uma vez instalado o pacote no R, o acesso aos manuais de ajuda do referido pacote, estarão disponíveis, graças a esses arquivos.
Instalação de um pacote
A instalação de um pacote via CRAN pode ser feito pela função install.packages(pkgs = "nome_pacote"). Por exemplo, vamos tentar instalar o pacote midrangeMCP, da seguinte forma:
install.packages("midrangeMCP")
Pode ser que nesse processo, dependendo de onde o usuário esteja executando essa linha de comando, interface do R ou RStudio, que seja solicitado o espelho por onde deseja fazer a instalação. Isso é apenas um atalho para ter um acesso mais rápido na instalação do pacote. A sugestão é escolher um espelho de seu país de origem.
Uma forma simples de se ter detalhes do pacote na internet, tais como, baixar o pacote fonte ou o pacote binário do midrangeMCP, por exemplo, é sempre usar essa url: http://cran.r-project.org/package=midrangeMCP. Para qualquer outro pacote, basta mudar o nome do pacote na url, e assim, estaremos na página do repositório do pacote. O pacote fonte, como falado anteriormente, é compactado com extensão .tar.gz, no caso, midrangeMCP_3.1.1.tar.gz. O pacote binário tem a compactação zipada, midrangeMCP_3.1.1.tar.zip para o Windows e midrangeMCP_3.1.1.tgz para o Macintosh. O acesso aos arquivos do pacote mencionados no esqueleto são disponíveis no pacote fonte.
Uma outra forma possível de instalação é baixar o arquivo do pacote fonte para o seu computador e instalá-lo,
install.packages(pkgs = "./midrangeMCP.tar.gz", repos = NULL, type = "source")
Consideramos que o arquivo do pacote esteja no diretório de trabalho do usuário. Caso contrário, deve ser informado o local onde pacote se encontra no computador. Para o Window ou Macintosh, é possível instalar também, a partir dos pacotes binários.
Muitos dos desenvolvedores, estão disponibilizando seus projetos de pacotes, principalmente no GitHub, inclusive com manuals de ajuda com maiores detalhes. Pode ser possível instalar esses pacotes por esse repositório. Precisamos inicialmente do pacote devtools, e posteriormente a instalação do pacote. Segue,
install.packages("devtools")
install_github("bendeivide/midrangeMCP")
Contudo, devemos dar a preferência pela instalação via CRAN. Por lá, teremos a garantia que os pacotes estão estáveis para a utilização nas referidas plataformas mencionadas acima.
Alguns pacotes, por falta de manutenção, seja por atualizações do R ou por qualquer outro motivo, podem se tornar incompatíveis para utilização sobre alguns dos três sistemas operacionais básicos (SO Windows, Unix e SO Mac) exigidos pelo R. Dessa forma, se as correções não forem feitas, estes pacotes e tornam órfãos, ou seja, desativados sob o CRAN. O primeiros pacotes sob o CRAN, por exemplo, não tinham o arquivo NAMESPACE, que hoje é exigido. Qualquer tentativa de instalação desses pacotes nessas situações, não serão bem sucedidas. Dessa forma, fizemos uma vídeo-aula, como tentativa de recuperar os pacotes desativados. Porém, deixemos claro que nem sempre é possível a instalação de pacotes desativados.
Objetivos de um pacote
A ideia de um pacote para um usuário R deve representar como uma ferramenta para otimizar suas atividades do dia-a-dia na utilização da linguagem. Suponha que o usuário seja um cientista de dados, e todos os dias ele carrega uma sequência de scripts, via source, para disponibilizar suas funções no ambiente global. Isso acaba gerando processos repetitivos de trabalho desnecessários.
Ao invés, o cientista de dados pode desenvolver um pacote, e esse pacote conter todas as funções necessárias para as suas análises. De uma vez, o pacote instalado e anexado no caminho de busca, todas as suas funções estarão disponíveis para utilização. Portanto, o entendimento disso, permite uma maior eficiência de trabalho.
Outro ponto é que a experiência contida em um pacote pode ser propagada mais facilmente para outros usuários, mostrando que o conhecimento é uma liberdade necessária. Tanto pelo CRAN, quanto por outras plataformas, o pacote pode ser disponibilizado.
Utilizar as funções de um pacote
Uma vez instalado o pacote, precisamos carregar e anexá-lo, para que possamos utilizar os recursos disponíveis no pacote, como funções, dados, etc. Isso significa, disponibilizar na memória e inseri-lo no caminho de busca, respectivamente. Para fazer essas duas ações ao mesmo tempo, use a função library() ou require(). A primeira função se for utilizada sem argumento algum, retorna todos os pacotes instalados na bibioteca de pacotes do R. Vejamos o exemplo do pacote midrangeMCP,
# Carregando e anexando o pacote midrangeMCP
library(midrangeMCP)
# Usando a função MRtest() desse pacote
#-----
# Dados simulados de um experimento em DIC (Delineamento Inteiramente Casualizado)
# Variavel resposta
rv <- c(100.08, 105.66, 97.64, 100.11, 102.60, 121.29, 100.80,
99.11, 104.43, 122.18, 119.49, 124.37, 123.19, 134.16,
125.67, 128.88, 148.07, 134.27, 151.53, 127.31)
# Tratamento
treat <- factor(rep(LETTERS[1:5], each = 4))
# Anava
res <- anova(aov(rv~treat))
DFerror <- res$Df[2]
MSerror <- res$`Mean Sq`[2]
# Aplicando testes
results <- midrangeMCP::MRtest(y = rv,
trt = treat,
dferror = DFerror,
mserror = MSerror,
alpha = 0.05,
main = "PCMs",
MCP = c("all"))
MCP's based on distributions of the studentized midrange and range
Study: PCMs
Summary:
Means std r Min Max
A 100.87 3.40 4 97.64 105.66
B 105.95 10.33 4 99.11 121.29
C 117.62 9.02 4 104.43 124.37
D 127.97 4.74 4 123.19 134.16
E 140.30 11.42 4 127.31 151.53
Mean Grouping Midrange Test
Statistics:
Exp.Mean CV MSerror Df n Stud.Midrange Ext.DMS Int.DMS
118.542 7.08182 70.47488 15 5 1.089968 5.90246 4.575105
Groups:
Means Groups
E 140.30 g1
D 127.97 g2
C 117.62 g3
B 105.95 g4
A 100.87 g4
Mean Grouping Range Test
Statistics:
Exp.Mean CV MSerror Df n Stud.Range DMS
118.542 7.08182 70.47488 15 5 4.366985 18.33027
Groups:
Means Groups
E 140.30 g1
D 127.97 g2
C 117.62 g2
B 105.95 g3
A 100.87 g3
SNK Midrange Test
Statistics:
Exp.Mean CV MSerror Df n Stud.Midrange DMS
comp1 118.542 7.0818 70.4749 15 5 1.0900 5.9025
comp2 118.542 7.0818 70.4749 15 4 1.1646 6.2159
comp3 118.542 7.0818 70.4749 15 3 1.2828 6.7121
comp4 118.542 7.0818 70.4749 15 2 1.5072 7.6536
Groups:
Means Groups
E 140.30 g1
D 127.97 g1
C 117.62 g1g2
B 105.95 g1g2
A 100.87 g2
Tukey Midrange Test
Statistics:
Exp.Mean CV MSerror Df n Stud.Midrange Ext.DMS Int.DMS
118.542 7.08182 70.47488 15 5 1.089968 5.90246 4.575105
Groups:
Means Groups
E 140.30 g1
D 127.97 g2
C 117.62 g3
B 105.95 g4
A 100.87 g4MCP's based on distributions of the studentized midrange and range
Study: PCMs
Summary:
Means std r Min Max
A 100.87 3.40 4 97.64 105.66
B 105.95 10.33 4 99.11 121.29
C 117.62 9.02 4 104.43 124.37
D 127.97 4.74 4 123.19 134.16
E 140.30 11.42 4 127.31 151.53
Mean Grouping Midrange Test
Statistics:
Exp.Mean CV MSerror Df n Stud.Midrange Ext.DMS Int.DMS
118.542 7.08182 70.47488 15 5 1.089968 5.90246 4.575105
Groups:
Means Groups
E 140.30 g1
D 127.97 g2
C 117.62 g3
B 105.95 g4
A 100.87 g4
Mean Grouping Range Test
Statistics:
Exp.Mean CV MSerror Df n Stud.Range DMS
118.542 7.08182 70.47488 15 5 4.366985 18.33027
Groups:
Means Groups
E 140.30 g1
D 127.97 g2
C 117.62 g2
B 105.95 g3
A 100.87 g3
SNK Midrange Test
Statistics:
Exp.Mean CV MSerror Df n Stud.Midrange DMS
comp1 118.542 7.0818 70.4749 15 5 1.0900 5.9025
comp2 118.542 7.0818 70.4749 15 4 1.1646 6.2159
comp3 118.542 7.0818 70.4749 15 3 1.2828 6.7121
comp4 118.542 7.0818 70.4749 15 2 1.5072 7.6536
Groups:
Means Groups
E 140.30 g1
D 127.97 g1
C 117.62 g1g2
B 105.95 g1g2
A 100.87 g2
Tukey Midrange Test
Statistics:
Exp.Mean CV MSerror Df n Stud.Midrange Ext.DMS Int.DMS
118.542 7.08182 70.47488 15 5 1.089968 5.90246 4.575105
Groups:
Means Groups
E 140.30 g1
D 127.97 g2
C 117.62 g3
B 105.95 g4
A 100.87 g4midrangeMCP::MRbarplot(results)
Carregando e anexando um pacote
Anteriormente, falamos que usamos a função library() ou require() para carregar e anexar um pacote para utilizar suas funções, após a instalação. Carregar um pacote significa disponibilizar na memória ativa. Para acessar uma função de um pacote após ter sido carregado, usamos o operador ::, isto é, nome_pacote::nome_função. Isto significa, que será chamado a função necessária sem anexar o pacote no caminho de busca. Estudaremos a seguir um pouco mais sobre caminho de busca. Para esse momento, entenda que é um caminho hierarquizado de ambientes, isto é, objetos que armazenam, em forma de lista, nomes associados a objetos. A função para ver o caminho de busca é search(). Em nosso caso, temos:
# Caminho de busca
search()
[1] ".GlobalEnv" "package:magrittr" "package:leaflet"
[4] "package:stats" "package:graphics" "package:grDevices"
[7] "package:utils" "package:datasets" "package:methods"
[10] "Autoloads" "package:base" # Carregando e chamando uma função de um pacote
midrangeMCP::MRwrite(results, extension = "latex")
Table in latex of results of the MGM test
% latex table generated in R 4.1.0 by xtable 1.8-4 package
% Tue Nov 09 13:21:55 2021
\begin{table}[ht]
\centering
\begin{tabular}{lrl}
\hline
trt & Means & Groups \\
\hline
E & 140.30 & g1 \\
D & 127.97 & g2 \\
C & 117.62 & g3 \\
B & 105.95 & g4 \\
A & 100.87 & g4 \\
\hline
\end{tabular}
\end{table}
Table in latex of results of the MGR test
% latex table generated in R 4.1.0 by xtable 1.8-4 package
% Tue Nov 09 13:21:55 2021
\begin{table}[ht]
\centering
\begin{tabular}{lrl}
\hline
trt & Means & Groups \\
\hline
E & 140.30 & g1 \\
D & 127.97 & g2 \\
C & 117.62 & g2 \\
B & 105.95 & g3 \\
A & 100.87 & g3 \\
\hline
\end{tabular}
\end{table}
Table in latex of results of the SNKM test
% latex table generated in R 4.1.0 by xtable 1.8-4 package
% Tue Nov 09 13:21:55 2021
\begin{table}[ht]
\centering
\begin{tabular}{lrl}
\hline
trt & Means & Groups \\
\hline
E & 140.30 & g1 \\
D & 127.97 & g1 \\
C & 117.62 & g1g2 \\
B & 105.95 & g1g2 \\
A & 100.87 & g2 \\
\hline
\end{tabular}
\end{table}
Table in latex of results of the TM test
% latex table generated in R 4.1.0 by xtable 1.8-4 package
% Tue Nov 09 13:21:55 2021
\begin{table}[ht]
\centering
\begin{tabular}{lrl}
\hline
trt & Means & Groups \\
\hline
E & 140.30 & g1 \\
D & 127.97 & g2 \\
C & 117.62 & g3 \\
B & 105.95 & g4 \\
A & 100.87 & g4 \\
\hline
\end{tabular}
\end{table}
Table in latex of results of descriptive statistics
% latex table generated in R 4.1.0 by xtable 1.8-4 package
% Tue Nov 09 13:21:55 2021
\begin{table}[ht]
\centering
\begin{tabular}{lrrrrr}
\hline
trt & Means & std & r & Min & Max \\
\hline
A & 100.87 & 3.40 & 4.00 & 97.64 & 105.66 \\
B & 105.95 & 10.33 & 4.00 & 99.11 & 121.29 \\
C & 117.62 & 9.02 & 4.00 & 104.43 & 124.37 \\
D & 127.97 & 4.74 & 4.00 & 123.19 & 134.16 \\
E & 140.30 & 11.42 & 4.00 & 127.31 & 151.53 \\
\hline
\end{tabular}
\end{table}
See yours tables in Console
Format: latex# Verificando novamente o caminho de busca
search()
[1] ".GlobalEnv" "package:magrittr" "package:leaflet"
[4] "package:stats" "package:graphics" "package:grDevices"
[7] "package:utils" "package:datasets" "package:methods"
[10] "Autoloads" "package:base" Com as linhas de comando apresentadas anteriormente, percebemos ao executar a função MRwrite() do pacote midrangeMCP, usando :: que o caminho de busca não foi alterado. Isso significa que o pacote não foi anexado, apenas carregado, ou seja, se o usuário desejar usar alguma função do pacote midrangeMCP digitando apenas o nome no console, não será possível, porque o pacote não está anexado ao caminho de busca.
Vejamos uma outra situação,
# Caminho de busca
search()
[1] ".GlobalEnv" "package:magrittr" "package:leaflet"
[4] "package:stats" "package:graphics" "package:grDevices"
[7] "package:utils" "package:datasets" "package:methods"
[10] "Autoloads" "package:base" # Carregando e anexando um pacote
library(midrangeMCP)
# Verificando novamente o caminho de busca
search()
[1] ".GlobalEnv" "package:midrangeMCP" "package:magrittr"
[4] "package:leaflet" "package:stats" "package:graphics"
[7] "package:grDevices" "package:utils" "package:datasets"
[10] "package:methods" "Autoloads" "package:base" # Chamando uma funcao do pacote
# guimidrangeMCP()
Com o uso da função library(), percebemos que o caminho de busca foi alterado, porque agora temos o ambiente de pacote package:midrangeMCP. Isso significa que agora poderemos acessar os objetos desse pacote apenas digitando o nome associado a eles. Por fim, a última linha foi comentada, porque é uma interface gráfica ao usuário para o pacote, o que chamamos de GUI (do inglês, Graphical User Interface), mas que pode ser visualizado na Figura 19.
.](images/midrangeMCP.png)
Figure 19: Interface para o pacote midrangeMCP.
NAMESPACE de um pacote
No início da seção sobre pacotes, falamos sobre o esqueleto de um pacote, isto é, os componentes básicos de um pacote. Um dos arquivos é o NAMESPACE. Esse arquivo é respondsável pela exportação e importação de funções. As funções exportadas de um pacote, por meio desse arquivo, são aquelas visíveis após a anexação do pacote ao caminho de busca, ou por meio do operador ::. As funções importadas são aquelas utilizadas de outros pacotes.
As funções ditas internas do pacote, são aquelas não mencionadas no NAMESPACE. Em muitas situações, precisamos de funções internas necessárias para a finalidade do pacote, que muitas vezes não é objetivo final para disponibilidade dos usuários, mas códigos intermediários para a boa funcionabilidade do pacote. Dessa forma, uma boa escolha para que não haja conflitos em nomes associados a objetos no ambiente de trabalho, é a decisão de não exportá-los.
Porém, quando se cria um pacote, por exemplo, pelo RStudio, o padrão no NAMESPACE é o comando:
exportPattern("^[^\\.]")que significa que todas as funções no pacote serão exportadas que não iniciam por um ponto (“.”). Como toda função em um pacote precisa de um arquivo de ajuda (.Rd), todas as funções deverão ter esses tipos arquivos inseridos no subdiretório man/.
Os primeiros pacotes submetidos ao CRAN na primeiras versões do R, não tinha o arquivo NAMESPACE. Por isso que quando tentamos instalar pacotes órfãs antigos, um dos erros é a falta desse arquivo.
Mais detalhes sobre o desenvolvimento de pacotes será abordado no módulo Programação em R (Nível Avançado).
Usando os operadores :: e :::
Como falamos anteriormente, para chamarmos uma função sem a necessidade de anexar o pacote, usamos o operador ::. Comentamos também, que algumas funções não eram exportadas pelo NAMESPACE de um pacote. Contudo, se desejarmos visualizar ou executá-las, poderemos utilizar o operador :::. Vejamos um exemplo,
# Instale o pacote SMR
# install.packages(SMR) # Descomente a linha de comando para instalar
# Carregando e chamando funcoes exportadas do pacote SMR
SMR::pSMR(q = 2, size = 10, df = 3)
[1] 0.9905216# Carregando e chamando funcoes nao exportadas ao pacote
SMR:::GaussLegendre(size = 4)
$nodes
[1] -0.8611363 -0.3399810 0.3399810 0.8611363
$weights
[1] 0.3478548 0.6521452 0.6521452 0.3478548As funções internas dos pacotes devem ser utilizadas com muita cautela, uma vez que são funções que podem passar por atualizações, mudanças. Isso porque, como não são funções exportadas, alguns pacotes podem passar por atualizações, e desse modo, estas funções também podem ser atualizadas ou até mesmo alteradas.
Outro ponto interessante é que não se recomenda a utilização de importação de funções internas de outros pacotes no desenvolvimento de pacotes, uma vez que são funções que podem passar por mudanças drásticas, e portanto, gerar problemas nas rotinas. Se uma função em um pacote não foi exportada, é porque o desenvolvedor tem um bom motivo para tal situação. As funções exportadas são de fato a essência do objetivo de um pacote, e por isso que elas são exportadas.
Ambientes e Caminho de busca
Na seção sobre funções, discutimos dois pontos interessantes sobre objetos, que é a atribuição e o escopo. E esses dois pontos estão intimamente relacionados ao objeto ambiente, de tipo "environment". O ambiente é um objeto que armazena, em forma de lista, as ligações dos nomes associados aos objetos. Porém, existem diferenças entre o objeto lista do objeto ambiente, com quatro exceções (Wickham (2019)):
- Cada nome deve ser único;
- Os nomes em um ambiente não são ordenados;
- Um ambiente tem um pai ou também chamado de ambiente superior;
- Ambientes não são copiados quando modificados.
Muitas dessas definições são complexas para esse momento. E uma profundidade sobre o assunto, será abordada no módulo Programação em R (Nível Intermediário). Contudo, introduziremos algumas características importantes para os objetos do tipo environment.
O ambiente de trabalho do R é conhecido como ambiente global, pois é onde todo o processo de interação da linguagem ocorre. Existe um nome específico associado a esse objeto, que é .GlobalEnv, ou também pode ser acessado pela função globalenv(). Para sabermos quais os nomes que existem nesse ambiente, usamos a função ls(), isto é,
# Nomes no ambiente global
ls()
character(0)Quando o resultado da função é character(0), significa que não existem nomes criados no ambiente global. O ambiente corrente de trabalho é informado pela função environment(), isto é,
# Comparando os ambientes
identical(environment(), .GlobalEnv)
[1] TRUE# Forma errada de comparar ambientes (Erro...)
environment() == .GlobalEnv
Error in environment() == .GlobalEnv: comparação (1) é possível apenas para tipos lista ou atômicosA segunda forma é equivocada, porque o operador booleano, ==, é aplicado apenas a vetores, e o objeto ambiente não é um vetor. Acrescentamos ainda que não podemos utilizar o sistema de indexação, isto é,
# Criando objetos no ambiente global
b <- 2; a <- "Ben"; x <- TRUE
# Verificando os nomes no ambiente global
ls()
[1] "a" "b" "x"# Acessando o objeto "a"
.GlobalEnv$a
[1] "Ben".GlobalEnv[["a"]]
[1] "Ben"# Acessando o primeiro nome (Erro...)
.GlobalEnv[[1]]
Error in .GlobalEnv[[1]]: argumentos errados para obtenção de subconjuntos de um ambienteA última linha de comando retorna um erro, porque os nomes em ambientes não são ordenados, ao invés, devemos chamar os resultados por meio de $ ou [[<nome_obj>]].
Poderemos criar um ambiente pela função new.env(), e inserir ligações dentro dele, como apresentados a seguir.
# Criando objetos no ambiente global
b <- 2; a <- "Ben"; x <- TRUE
# Verificando os nomes no ambiente global
ls()
[1] "a" "b" "x"# Criando um objeto ambiente no ambiente global
amb1 <- new.env()
# Inserindo nomes nesse no ambiente "amb1"
amb1$d <- 3; amb1$e <- "FALSE"
# Verificando nomes no ambiente global
ls()
[1] "a" "amb1" "b" "x" # Verificando nomes no ambiente "amb1"
ls(envir = amb1)
[1] "d" "e"Todo ambiente tem um ambiente pai ou ambiente superior. Quando um nome não é encontrado no ambiente corrente, o R procurará no ambiente pai. Para saber, use parent.env(), isto é,
parent.env(amb1)
<environment: R_GlobalEnv>O único ambiente que não tem pai é o ambiente vazio, objeto emptyenv(), que pode ser observado pela linha de comando:
Error in parent.env(emptyenv()): o ambiente vazio não tem paiA superatribuição <<-
A atribuição (<-) é uma função que associa um nome a um objeto no ambiente corrente. Quando usamos o R, quase sempre esse ambiente é o ambiente global. A superatribuição (<<-) cria um nome e o associa a um objeto no ambiente pai do ambiente de onde essa associação está sendo criada. Vejamos,
# Criando o objeto x e o imprimindo
x <- 0; x
[1] 0# Criando uma funcao com a superatribuicao
f1 <- function() {
# Obj2
x <- 1
# Modificando x do ambiente global
x <<- 2
# Imprimindo o ambiente de execucao
env <- environment()
# Imprimindo o Obj2
res <- list(x = x, "Ambiente de execução" = env, "Ambiente Pai" = parent.env(env))
# Retornando a lista
return(res)
}
# Imprimindo f1
f1()
$x
[1] 1
$`Ambiente de execução`
<environment: 0x0000000009b9a698>
$`Ambiente Pai`
<environment: R_GlobalEnv># Imprimindo x
x
[1] 2# Imprimindo o ambiente envolvente de f1
environment(f1)
<environment: R_GlobalEnv># Imprimindo os nomes do ambiente global
ls()
[1] "a" "amb1" "b" "cran" "f1" "github"
[7] "rlink" "rstudio" "x" Esse caso é interessante porque vemos dois nomes associados a objetos em ambientes diferentes. Alguns ambientes são criados pela função function(), são os chamados ambientes funcionais. Um deles é o ambiente envolvente, já comentado na seção sobre funções. O ambiente envolvente da função f1 é o ambiente global. Já no corpo da função f1, um outro ambiente surge quando a função é chamada, é o ambiente de execução. Toda vez que a função é chamada, cria-se um novo ambiente de execução. Observemos os identificadores, em Ambiente de execução, quando executamos a função mais de uma vez,
f1()$`Ambiente de execução`
<environment: 0x0000000028ea9b38>f1()$`Ambiente de execução`
<environment: 0x000000002b2f74d8>f1()$`Ambiente de execução`
<environment: 0x000000002b04bd60>O ambiente pai do ambiente de execução, é o ambiente envolvente de f1, que nesse caso é o ambiente global. Assim, observe que o ocorre quando executamos o comando de superatribuição. O nome x no ambiente global passou a está associado ao valor 2, porque foi alterado por <<-, mas o nome x continuou associado ao valor 1, porque a função f1() retornou o valor 1. Isso mostra que a superatribuição não cria um objeto no ambiente atual, mas em um ambiente pai se não existe ou altera o nome existente. Vejamos o complemento dessa afirmação no próximo exemplo.
# Verificando os nomes no ambiente global
ls()
character(0)# Criando uma funcao
f2 <- function() {
x <<- 2
}
# Executando f2
f2()
# Verificando novamente os nomes no ambiente global
ls()
[1] "f2" "x" # Verificando o valor de x
x
[1] 2Com a superatribuição executada dentro de f2() e como no ambiente pai não eistia o nome x, este foi criado e associado ao valor 2. Um próximo exemplo, consideramos um ambiente envolvente que não seja o ambiente global. Vejamos,
# Funcao contador
contador <- function() {
i <- 0
env1 <- environment()
aux <- function() {
# do something useful, then ...
i <<- i + 1
env2 <- environment()
res2 <- list(i = i, `AmbExec_aux` = env2, `AmbExec_contador` = env1)
return(res2)
}
}
# Chamada de funcao
contador1 <- contador()
contador1()
$i
[1] 1
$AmbExec_aux
<environment: 0x000000002ae7d6e0>
$AmbExec_contador
<environment: 0x000000002af0ea08>contador1()
$i
[1] 2
$AmbExec_aux
<environment: 0x000000002adca7d0>
$AmbExec_contador
<environment: 0x000000002af0ea08>contador1()
$i
[1] 3
$AmbExec_aux
<environment: 0x000000002ad1b2d8>
$AmbExec_contador
<environment: 0x000000002af0ea08># Chamada de funcao
contador2 <- contador()
contador2()
$i
[1] 1
$AmbExec_aux
<environment: 0x000000000647ce58>
$AmbExec_contador
<environment: 0x000000002ab3add8>Quando uma função function() é criada dentro de outra função function() o ambiente de execução da função superior, contador(), é o ambiente envolvente da função interna, aux(). Dessa forma, o ambiente de execução de contador() não será mais efêmero, isto é, não será apagado após a execução, como pode ser visto em contador1(). Observamos que executamos contador1() três vezes. O nome i foi atualizado, devido a superatribuição, a cada chamada da mesma função. Ao passo que, quando realizamos uma nova chamada de contador(), por meio de contador2(), o resultado de i retora o valor 1, porque um novo ambiente de execução para contador() foi criado, como pode ser observado.
Os demais ambiente funcionais e exemplos, serão descritos no módulo Programação em R (Nível Intermediário).
Caminho de busca
Por fim, uma última forma do R encontrar os nomes é pelo caminho de busca, que além dos ambientes criados e o ambiente global, existem os ambientes de pacotes. Toda vez que um pacote é anexado ao caminho de busca, o ambiente de pacote anexado será sempre o pai do ambiente global. Vejamos,
# Caminho de busca
search()
[1] ".GlobalEnv" "package:magrittr" "package:leaflet"
[4] "package:stats" "package:graphics" "package:grDevices"
[7] "package:utils" "package:datasets" "package:methods"
[10] "Autoloads" "package:base" [1] ".GlobalEnv" "package:SMR" "package:magrittr"
[4] "package:leaflet" "package:stats" "package:graphics"
[7] "package:grDevices" "package:utils" "package:datasets"
[10] "package:methods" "Autoloads" "package:base" # Carregando o pacote midrangeMCP
library(midrangeMCP)
# Verificando o caminho de busca
search()
[1] ".GlobalEnv" "package:midrangeMCP" "package:SMR"
[4] "package:magrittr" "package:leaflet" "package:stats"
[7] "package:graphics" "package:grDevices" "package:utils"
[10] "package:datasets" "package:methods" "Autoloads"
[13] "package:base" A lista dos ambientes no caminho de busca segue a ordem hierárquica dos ambientes, de modo que o ambiente global será sempre o ambiente de trabalho, isto é, o ambiente corrente. Não foi apresentado nessa lista, o ambiente vazio, emptyenv(). Mas, poderemos utilizar o pacote rlang para isso.
# Criando um ambiente
amb2 <- new.env()
# Verificando seus parentais
rlang::env_parents(env = amb2, last = emptyenv())
[[1]] $ <env: global>
[[2]] $ <env: package:midrangeMCP>
[[3]] $ <env: package:SMR>
[[4]] $ <env: package:magrittr>
[[5]] $ <env: package:leaflet>
[[6]] $ <env: package:stats>
[[7]] $ <env: package:graphics>
[[8]] $ <env: package:grDevices>
[[9]] $ <env: package:utils>
[[10]] $ <env: package:datasets>
[[11]] $ <env: package:methods>
[[12]] $ <env: Autoloads>
[[13]] $ <env: package:base>
[[14]] $ <env: empty>Dessa forma, é caminho de busca que o R procurará pelos nomes. Isso significa, que se o ambiente envolvente de uma função, por exemplo, for o ambiente vazio, o R procurará pelas funções básicas no pacote base e não será encontrado, pois nesse ambiente não há nomes e nem ambientes parentais. Por isso, que o R depende do escopo léxico para tudo. Vejamos,
# Criando uma funcao
f3 <- function() x + 1
# Modificando o ambiente envolvente de f3
environment(f3) <- emptyenv()
# Dependencias externas da funcao f3
codetools::findGlobals(f3)
[1] "+" "x"# Chamando a funcao f3
f3()
Error in x + 1: não foi possível encontrar a função "+"Isso não ocorre quando definimos o ambiente envolvente de f3() como sendo o ambiente global, porque quando a função buscar pelo operador de soma neste ambiente e não encontrar, f3() seguirá até o ambiente package:base, para encontrar o operador “+.”
Por isso, que usar a função attach() para anexar objetos do tipo quadro de dados (data frames), por exemplo, pode se tornar um problema em um código, quando temos nomes iguais para objetos diferentes. Para isso, apresentamos o código a seguir.
# objeto quadro de dados
dados <- data.frame(sd = 1:3, var = (1:3)^2)
# Caminho de busca
search()
[1] ".GlobalEnv" "package:midrangeMCP" "package:SMR"
[4] "package:magrittr" "package:leaflet" "package:stats"
[7] "package:graphics" "package:grDevices" "package:utils"
[10] "package:datasets" "package:methods" "Autoloads"
[13] "package:base" # anexando "dados" ao caminho de busca
attach(dados)
# Verificando novamente o caminho de busca
search()
[1] ".GlobalEnv" "dados" "package:midrangeMCP"
[4] "package:SMR" "package:magrittr" "package:leaflet"
[7] "package:stats" "package:graphics" "package:grDevices"
[10] "package:utils" "package:datasets" "package:methods"
[13] "Autoloads" "package:base" # Imprimindo sd
sd
[1] 1 2 3# Desanexando "dados"
detach(dados)
# Imprimindo sd
sd
function (x, na.rm = FALSE)
sqrt(var(if (is.vector(x) || is.factor(x)) x else as.double(x),
na.rm = na.rm))
<bytecode: 0x0000000029010c70>
<environment: namespace:stats>Quando criamos o objeto dados, uma de suas colunas estava nomeada por sd, que também é o nome de uma função do pacote stats, que representa a variância. Porém, quando anexamos o objeto dados no caminho de busca, um novo ambiente é criado, como pai do ambiente global e com mesmo nome do objeto, e os elementos do objeto dados são copiados para esse ambiente. Assim, o nome sd foi procurado e não encontrado, seguindo a busca para o próximo ambiente que foi dados, e daí foi encontrado. Percebemos que também existe esse nome no ambiente do pacote stats, que é do tipo função. Entretanto, como o primeiro objeto encontrado associado a esse nome estava no ambiente dados, ele é retornado. Nesses casos, se usarmos a superatribuição, a alteração ocorrerá apenas na cópia dos elementos no ambiente anexado, e não nos elementos do objeto original. Caso haja a atribuição, o nome será criado no ambiente global.
Isso pode acabar se tornando um problema se muitos objetos forem anexados. Por isso, é preferível o uso da indexação ou $ para acessar os elementos de uma lista ou quadro de dados, evitando assim, conflitos na procura de nomes.
Desse mesmo modo, poderíamos pensar que esses mesmo conflitos poderiam surgir dentro de um pacote. Porém, graças ao NAMESPACE, isso não ocorre, sendo um dos assuntos abordados nos próximos módulos.
Interfaces com outras linguagens
Como afirmado no terceiro princício,
- Princípio da Interface: Interfaces para outros programas são parte do
R,
na base do R temos integrações prontas para implementar códigos em C e FORTRAN, como também outras linguagens. Por exemplo, na base do R existe um pacote chamado tcltk que integra a linguagem Tcl/Tk para o R. Esse pacote nos permite desenvolver interfaces gráficas para os nossos códigos.
Diversos outros pacotes, disponibilizados sob o CRAN realizaram diversas outras linguagens, que elencamos alguns,
| Pacote | Linguagem integrada |
|---|---|
tcltk |
Linguagem Tcl/Tk |
| RGtk2 | Linguagem Gtk+ |
| rJava | Linguagem Java |
| rmarkdown | Linguagens HTML, JavaScript, CSS, Markdown, \(\LaTeX\) |
| reticulate | Linguagem Python |
| JuliaCall, XRJulia | Linguagem Julia |
| Rcpp | Linguagem C++ |
| gecoder | Linguagem Ruby |
Existe um aplicativo da web bem interessante que integra as linguagem Julia, Python e R, chamado Jupyter. Muitas outras interfaces são integradas ao R. Ao longo do que formos pesquisando sobre mais pacotes, vamos atualizando a tabela.
Como motivação para esse momento, iremos realizar uma aplicação com as linguagens Python, C++ e Tcl/tk, para verificarmos que essas integrações são fáceis de serem realizadas. Claro, em algumas outras situações, como a integração do código C para R usando as funções .C() ou .Call() exigem um pouco mais de conhecimento. Contudo, no módulo Programação em R (Nível Avançado), iremos realizar algumas integrações com um abordagem mais profunda para algumas linguagens.
Implementação em Python
Vamos inicialmente instalar o pacote reticulate,
install.packages(reticulate)
Existem diversas formas de integração. Vamos pensar na forma mais básica, isto é, temos um script Python, isto é, um arquivo com a extensão .py, e vamos carregá-lo no R. O script, chamaremos de add.py, que segue:
def add(x, y):
return x + yVamos agora chamar a função add() em um script R:
# Carregando o script add.py
reticulate::source_python('add.py')
# Chamando a funcao add
add(5, 10)
[1] 15Implementação em C++
Vamos instalar o pacote Rcpp,
install.packages(Rcpp)
Vamos realizar a mesma aplicação feita em Python, que segue o script C++, com extensão .cpp,
#include <Rcpp.h>
using namespace Rcpp;
// [[Rcpp::export]]
int add(int x, int y) {
return x + y;
}Vamos agora chamar a função add() em um script R:
Implementação em TclTk
Nesse caso, não precisaremos instalar pacotes, pois o pacote tcltk é da Base do R. Ao invés, anexe-o no caminho de busca, use library(tcltk). Iremos implementar uma interface gráfica contendo uma janela com entradas de dois números e um botão para calcular a soma. Segue o script,
# Anexando o pacote
library(tcltk)
# Janela principal
main <- tktoplevel(width = 400, height = 300)
tkpack.propagate(main, FALSE)
# Texto inicial
tkpack(tklabel(main, text = "Soma de dois números inteiros"))
# Quadro 1
tkpack(q1 <- tkframe(main), side = "top")
tkpack(tklabel(q1, text = "Insira o primeiro número: ", padx = 3), side = "left", anchor = "e")
# Entrada 1
var1 <- tclVar("Insira um número inteiro")
tkpack(entry1 <- tkentry(q1, textvariable = var1, width = 25), side = "left", anchor = "ne")
# Quadro 2
tkpack(q2 <- tkframe(main), side = "top")
tkpack(tklabel(q2, text = "Insira o segundo número: ", padx = 3), side = "left", anchor = "e")
# Entrada 2
var2 <- tclVar("Insira um número inteiro")
tkpack(entry2 <- tkentry(q2, textvariable = var2, width = 25), side = "left", anchor = "ne")
# Botao
tkpack(botao <- tkbutton(main, text = "Somar dois números"), side = "top")
# Funcao auxiliar
f1 <- function(...) {
if ((is.na(as.numeric(tclvalue(var1))) | is.na(as.numeric(tclvalue(var2))))) {
tkpack(tklabel(main, text = "Insira Valores numéricos!"), side = "top")
} else {
res <- as.numeric(tclvalue(var1)) + as.numeric(tclvalue(var2))
tkpack(tklabel(main, text = paste("A soma é igual a ", res)), side = "top")
}
}
# Acao ao Botao
tkbind(botao, "<ButtonRelease>", f1)
O vídeo abaixo apresenta o resultado da interface.
Existem outras formas de chamar as rotinas em Python, C++ e Tcl/Tk no R, implementadas nos pacotes reticulate, Rcpp e tcltk, respecitvamente. Contudo, iremos apresentá-los no módulo Programação em R (Nível Avançado), bem como a integração de outras linguagens.
Considerações e preparação para o módulo Programação em R (Nível Intermediário)
Nesse módulo, tentamos tratar as ideias básicas por trás dos três princípios do R, mas principalmente os fundamentos de um objeto e a ideia sobre função. Tentamos repassar algo mais técnico sobre esta linguagem. Contudo, sabemos que algo introdutório deve ter uma flexibilidade quanto a profundidade das discussões e exemplos abordados. Mesmo assim, esse módulo dará suporte para problemas mais complexos que abordaremos para os módulos seguintes.
Faremos constantemente atualizações, quando necessárias, para melhorar esse material, e assim, auxiliar nos estudos de quem desejar entrar no mundo da linguagem R. Para isso, pedimos também a sua contribuição como leitor. Caso encontre alguma discordância nesse material ou complementos sobre a discussão, entre em contato pelas nossas redes sociais, como também por email: ben.deivide@ufsj.edu.br. Será muito bom podermos compartilhar as nossas experiências, uma vez que o conhecimento é uma liberdade necessária.
Na Figura 20, apresentamos um resumo do que vem para o módulo Programação em R (Nível Intermediário).
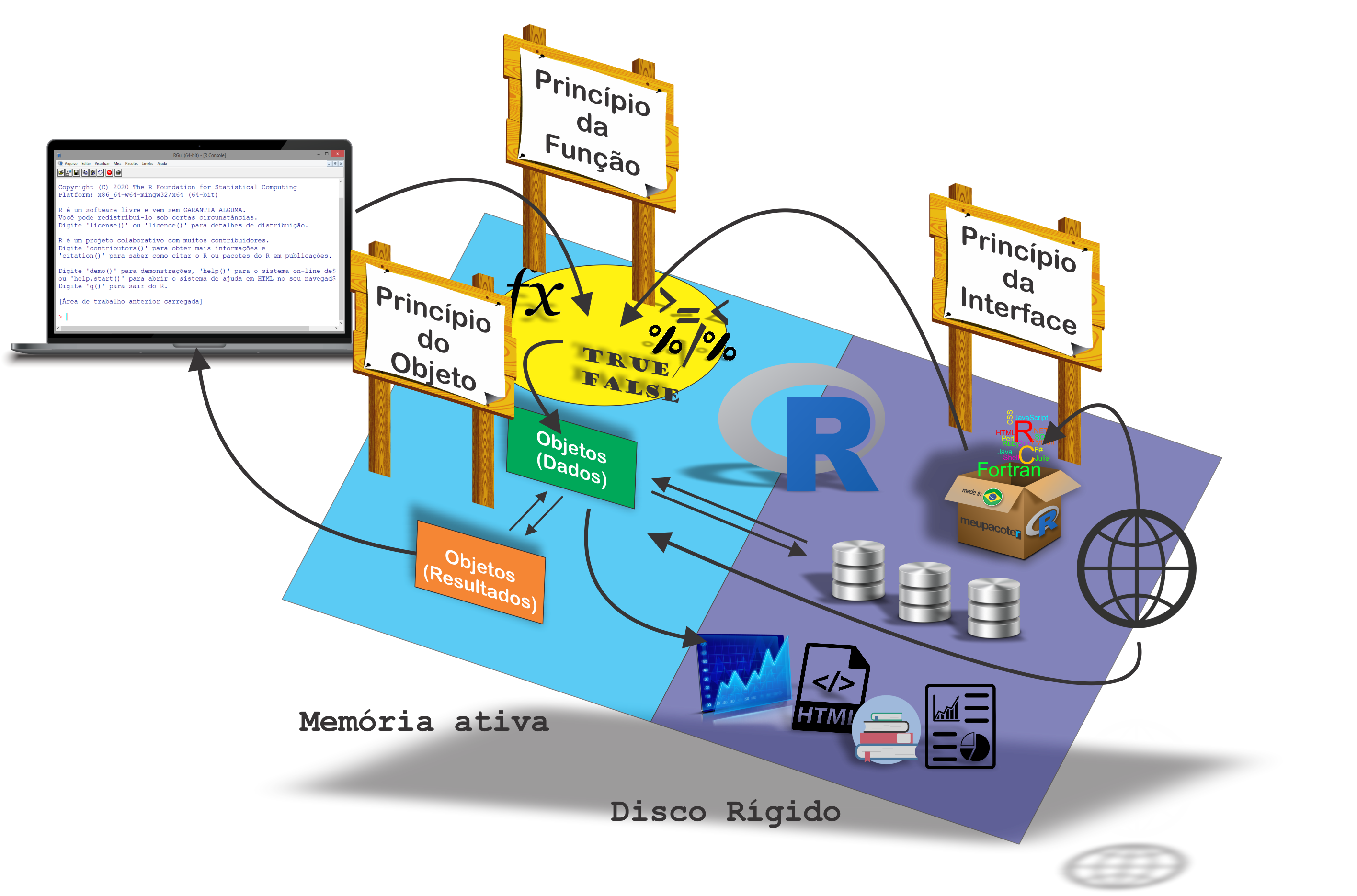
Figure 20: Relembrando como o R funciona.
Dentre os assuntos abordados, teremos:
- Aprofundamento sobre os objetos, associando-os com a tipagem da linguagem C;
- Cópias de objetos;
- Aprofundamento sobre Ambientes;
- Programação Orientada a objetos, bem como estudo de Métodos e Classes;
- Programação Funcional:
- Evitando loops;
- Eficiência;
- Programando em baixo nível;
- Programação defensiva;
- Introdução a Criação de Pacotes;
- Gráficos;
- Criação de documentos estáticas e dinâmicas;
Dessa forma, agradecemos imensamente o interesse como leitor, e também lhe provoco para participar juntamente conosco nessa propagação de conhecimento. Até o próximo módulo!
Fonte das fotos: Robert Gentleman do site: https://biocasia2020.bioconductor.org/ e Ross Ihaka do site: https://www.stat.auckland.ac.nz/en/about/news-and-events-5/news/news-2017/2017/12/ross-ihaka-retires.html↩︎
Fontes: https://cran.r-project.org/doc/html/interface98-paper/paper_2.html e https://www.r-project.org/contributors.html↩︎
Fonte: https://stat.ethz.ch/pipermail/r-announce/1997/000001.html↩︎
Fonte: https://www.gnu.org/philosophy/open-source-misses-the-point.html↩︎
Fonte: https://www.gnu.org/philosophy/open-source-misses-the-point.html↩︎
https://cran.r-project.org/doc/html/interface98-paper/paper_1.html↩︎
https://cran.r-project.org/doc/html/interface98-paper/paper_1.html↩︎
Fonte da foto: https://rstudio.com/speakers/j.j.-allaire/↩︎
Do ingles, Integrated Development Environment, que significa Ambiente de Desenvolvimento Integrado, como por exemplo, o RStudio, Emacs, dentre outros, para o
R.↩︎Comprehensive R Archive Network↩︎
Fonte da foto: https://www.ufpr.br/portalufpr/noticias/disciplinas-transversais-para-programas-de-pos-graduacao-abrem-inscricoes-nesta-segunda-feira/↩︎
Fonte da foto: Retirada de sua página pessoal, https://statweb.stanford.edu/~jmc4/↩︎
Basta usar a função
invisible(10 + 15), que a expressão é avaliada mas não impressa↩︎ou também
double, usando a funçãotypeof()↩︎sinônimo: string, cadeia de caracteres.↩︎
Diferente de outras linguagens, como a
C, que o contador começa do número 0.↩︎Para esse caso, use
mode()para verificar.↩︎A anexação de um pacote no caminho de busca pressupõe que este esteja instalado no seu computador↩︎
Use a função
typeof()para verificar.↩︎Use a função
typeof()para verificar.↩︎Use
typeof()para verificar o modo.↩︎
